Blog
PFNリサーチャーの武本です。この投稿は2025年12月19日にCommunications Chemistry誌に出版された論文「Generalizable Compound–Protein Interaction Prediction with a Model Incorporating Protein Structure–Aware and Compound Property–Aware Language Model Representations」についての解説記事です。論文はオープンアクセスで以下のURLから閲覧可能です。 https://www.nature.com/articles/s42004-025-01844-0
概要
創薬やケミカルバイオロジーの分野において、標的タンパク質と化合物の結合親和性を予測するCPI (Compound–Protein Interaction) 予測は、重要な役割を担います。しかし、既存の機械学習ベース手法の多くは、タンパク質配列やSMILESのような1次元情報に依存しており、タンパク質の立体構造や化合物の物性情報といった多角的な情報を十分に活用できていませんでした。その結果、学習データに含まれない新規ターゲットや新規化合物に対する汎化性能の低下という課題が指摘されていました。
これらの課題に対し、我々はCPI予測モデルGenSPARC (Generalized Structure- and Property-Aware Representations for CPI prediction) を開発しました。GenSPARCは、タンパク質と言語モデル、配列、構造、物性といった複数モダリティを統合することで、未知のターゲット・化合物に対しても高い予測性能を達成することを目指したモデルです。GenSPARCの主な技術的特徴は以下の通りです。
- 構造認識タンパク質言語モデル SaProt の活用: AlphaFold2で予測したタンパク質構造からFoldseekによる3Di構造アルファベットを付与し、「配列+構造」を同時に扱うStructure-Awareな表現を利用
- GCNと化合物言語モデルSPMMによる化合物表現: 分子グラフに対するグラフ畳み込みニューラルネットワーク (Graph Convolutional Neural Network; GCN) と、RDKitベースの物性記述子を入力とするproperty encoderを組み合わせ、構造と物性の両面から化合物の表現を取得
- マルチモーダル注意機構 (MAN) による残基・原子間の相互作用モデリング: タンパク質残基と化合物原子の間でCross-Attentionを実施し、結合親和性予測だけでなく、残基–原子レベルのコンタクトマップ予測も同時に行う
Karimi, Davis, KIBA, Metzといった標準的なCPI予測ベンチマークに加え、DUD-Eを用いた仮想スクリーニング設定で評価した結果、GenSPARCは既存の配列ベース・構造ベースの手法を上回る性能を示しました。特に、学習データに存在しないタンパク質・化合物(Unseen-Both)の組み合わせや、実験構造がない状況(AlphaFold2の予測構造のみ使用可能)においても、安定した高い予測性能を維持できることが最大の特徴です。
本稿では、GenSPARCのモデル構成、学習方法、ベンチマーク結果、および創薬実務における活用方向性について、技術的な観点から解説します。
背景
既存の CPI 予測の手法と課題
広く用いられているCPI予測手法は、大きく分けて「原子レベルの3次元構造を直接扱うアプローチ」と「配列・グラフ情報に基づく深層学習アプローチ」に分類されます。
「原子レベルの3次元構造を直接扱うアプローチ」としてはドッキングシミュレーションやMDシミュレーション、MDを利用した自由エネルギー計算法が挙げられます。これらの手法は物理的に解釈が容易であり、単一のターゲットに対して高精度な評価が可能であるという利点があります。しかしながら、以下のような課題も知られています。
- 信頼できるタンパク質構造(通常は実験構造)を事前に準備する必要がある
- 多数の候補化合物に対して適用するには計算コストが高い
- AlphaFold などの予測構造をそのまま用いると、ポケット形状や側鎖の原子配置のわずかな誤差により、性能が低下しやすい
一方、深層学習技術の発展に伴い、タンパク質の3次元原子配置を直接用いない「配列・グラフ情報に基づく深層学習アプローチ」に基づくモデル (DeepAffinity[1], GraphDTA[2], TransformerCPI[3]など) が複数提案されています。Fig. 1にそれらに共通する構造の概念図を示しています。これらのモデルは、実験構造が存在しなくても CPI予測が可能であるという利点がありますが、以下の問題点が指摘されています。
- 学習に使えるデータが限定されている
- タンパク質の立体構造や化合物の物性といった情報を十分に活用できていない
- 訓練に用いたデータから大きく乖離すると精度が低下するため、新規のターゲット・化合物に対する汎化性が低い
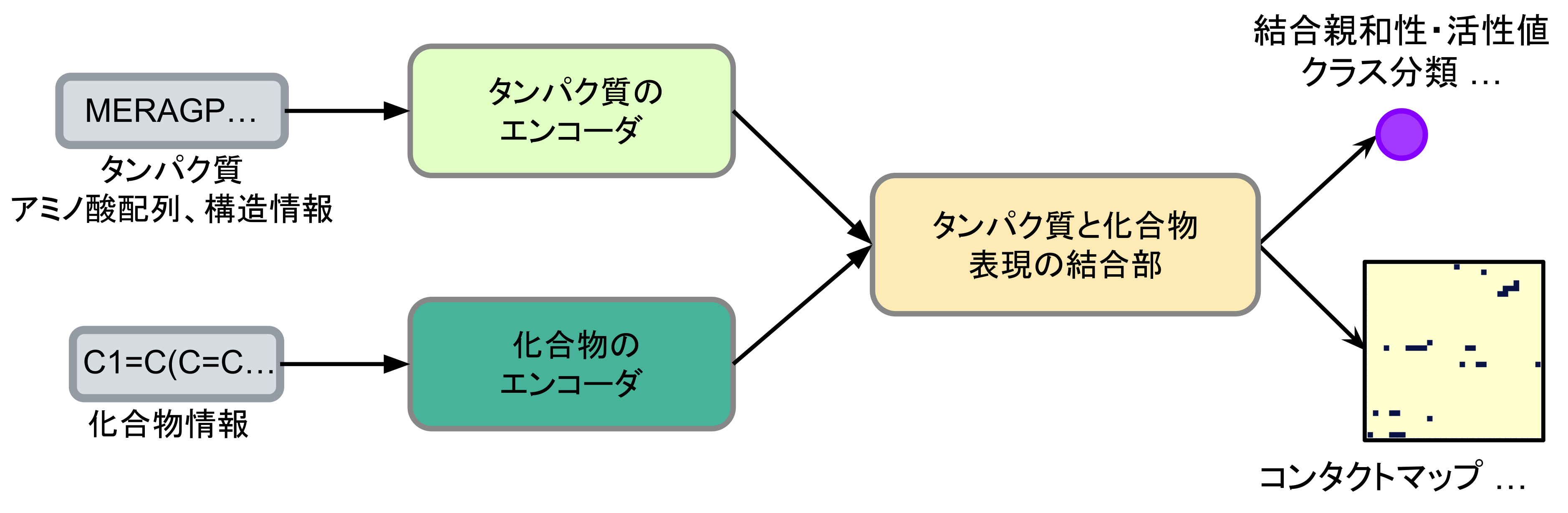
Fig. 1 深層学習を利用したCPI予測モデルの概念図
pLM と Structure-Aware pLM の登場
自然言語処理の成功を受け、アミノ酸配列を単語列とみなすタンパク質言語モデル (pLM) が開発されてきました (ESM[4] ファミリーなど)。しかし最近の研究では、pLMにおいてスケーリング則の破綻、すなわちパラメータ数を増やしても性能が頭打ちになる現象が観測されています[5]。そこで、配列だけでなく 構造情報も同時に学習させるpLM、 つまりStructure-Aware pLM が提案されました。例えば SaProt[6] では、AlphaFold2 で予測した構造から Foldseek[7]の3Di 構造アルファベットを割り当て、「アミノ酸文字+構造文字」からなるシーケンスとしてTransformerに学習させています。この文字列としての構造表現は、原子座標の細かい揺れに頑健であり、従来のpLMの限界を超える有望な方向性として注目されています。
方法
GenSPARC のアイデア:構造認識 pLM × 化合物言語モデル
GenSPARCは、このStructure-Aware pLMの利点と、化合物の言語モデルを組み合わせたマルチモーダルなCPI予測モデルです。全体像をFig. 2に示します。
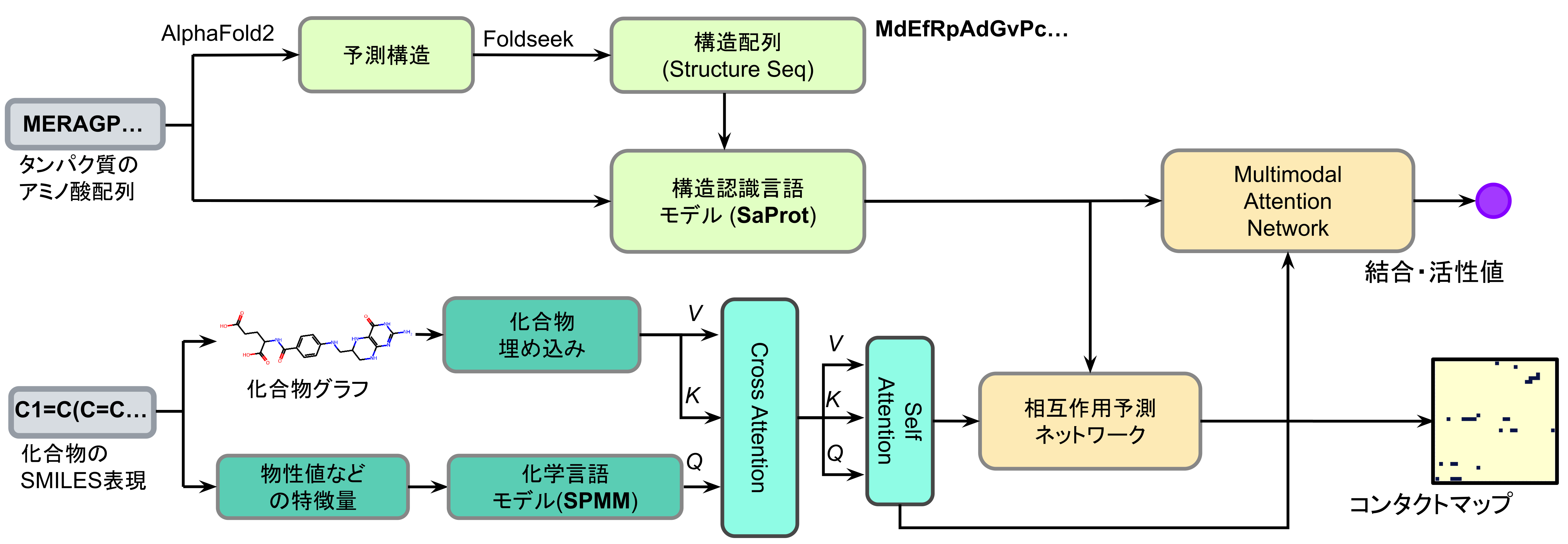
Fig. 2 GenSPARC モデルアーキテクチャの全体像
1. タンパク質エンコーダ:SaProt による構造認識ベクトル
各残基の埋め込みベクトルに配列と立体構造の両方を反映した表現を獲得します
- タンパク質配列から AlphaFold2 で構造を予測
- Foldseekで各残基に 3Di 構造アルファベットを割り当てる
- 「アミノ酸文字+構造文字」からなるstructure sequenceをSaProt に入力
- SaProt の出力ベクトルを線形変換し、GenSPARC用のタンパク質埋め込みにする
2. 化合物エンコーダ:GCN + SPMM による構造と物性の統合
化合物については、トポロジカルな構造と物理化学的な物性値の2種類の情報を使用します。
- 分子グラフ (原子と結合)
- RDKit で SMILES からグラフを作成
- 原子種、芳香族性、荷電状態などを特徴量にしたノードを GCN (3層) で処理
- 53 個の分子記述子
- 化合物言語モデルSPMM[8]のproperty encoderを利用
- aromaticity, Gasteiger charge, ring 数など RDKit ベースの物性を入力し、768 次元ベクトルを出力
これら2つの出力をマルチヘッドアテンションを用いたMolecular Fusionモジュールで統合し、「構造と物性の双方の情報を取り込んだ化合物ベクトル」を構築します。
3. マルチモーダル注意機構 (MAN)
タンパク質埋め込みと化合物埋め込みは、Multimodal Attention Network (MAN) (Fig. 3)で統合されます。
- Self-Attention:タンパク質内/化合物内の文脈をとらえる
- Cross-Attention:残基クエリが化合物キーを、原子クエリがタンパク質キーを参照し、残基と原子が互いを参照しながら特徴を更新(相互作用モデリング)
更新されたベクトルを平均プーリングした後、MLPで結合親和性 (回帰タスク) を予測します。さらに、残基ベクトルと原子ベクトルの内積からコンタクトマップ (残基-原子間の相互作用) も同時に予測します。
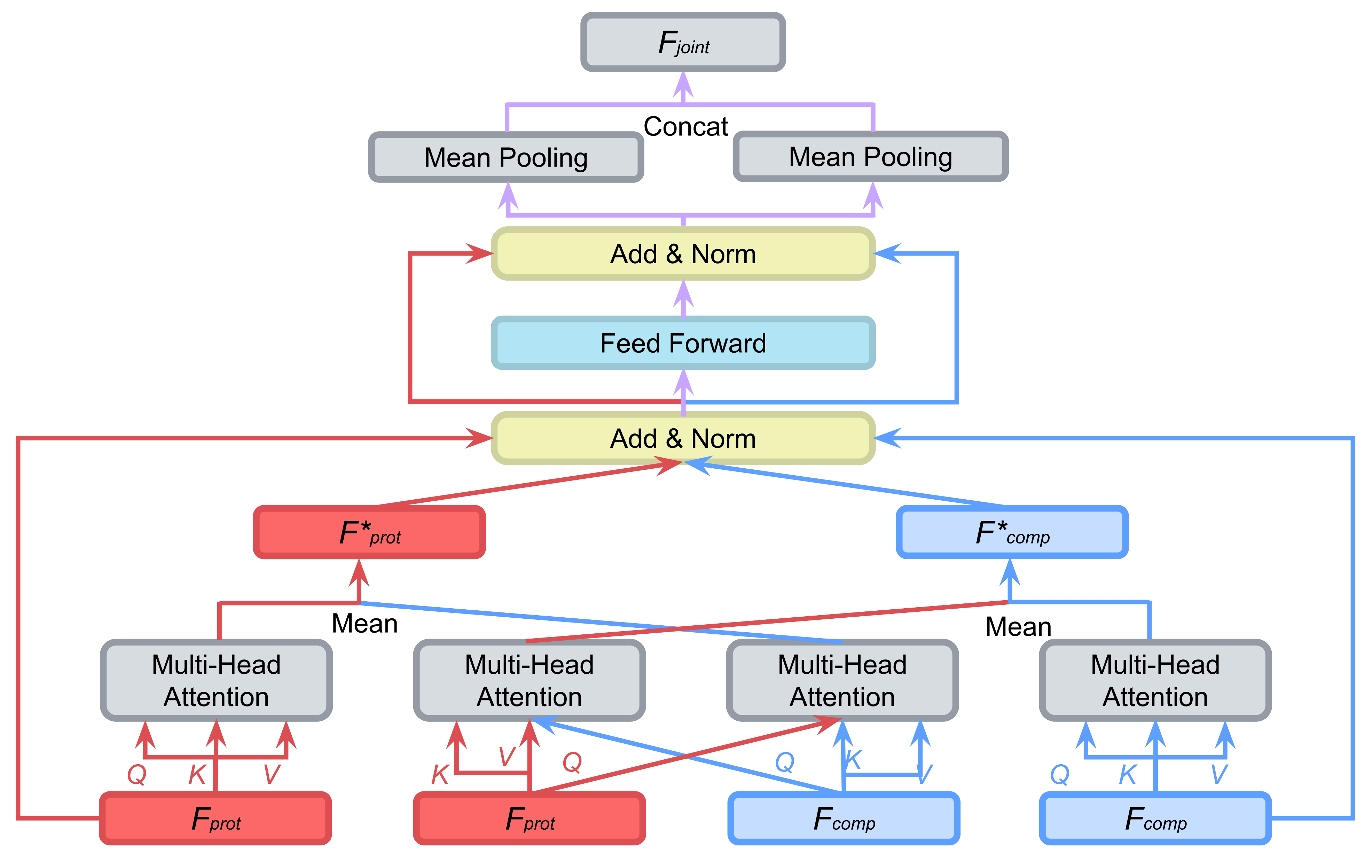
Fig.3 Multimodal Attention Network (MAN) の内部構造
結果
Karimiデータセットでのコンタクト & 親和性予測
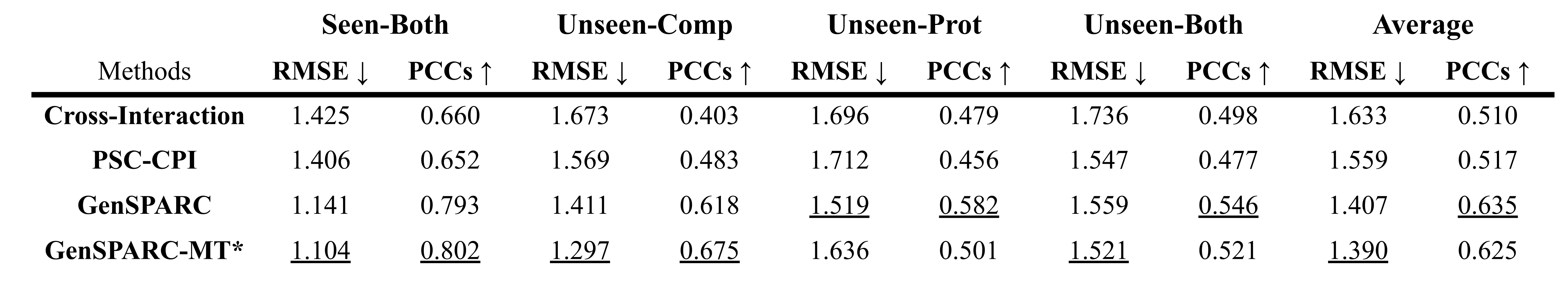
CPIの代表的ベンチマークであるKarimiデータセット[9]に対し、実用を見据えた類似度に基づく厳密なデータ分割 (Sequence-hard/Structure-hard) を再定義し評価を行いました。化合物はTanimoto類似度0.3以下、タンパク質はFoldseekによる構造配列一致度0.5以下をUnseenと定義するStructure-hard設定における評価結果の抜粋を表1(コンタクトマップ予測)および表2(結合親和性予測)に示します。なお表中のGenSPARC-MTはコンタクト予測と結合親和性回帰を同時に予測するマルチタスク (MT) 学習を行ったものです。
- コンタクトマップ予測 (表1):Structure-hard設定のUnseen-Bothなど、汎化性能が厳しく問われる設定においても、GenSPARCは既存手法(Cross-Interaction[10], PSC-CPI[11]など)を大きく上回るPRC-AUC / ROC-AUCを達成。
- 結合親和性予測 (表2):Unseen-Bothを含む全ての分割で、既存手法に比べて良好な性能を示した
このほか、キナーゼ中心のベンチマークデータセット Davis/KIBA/Metzの各データセットでも、Unseen both splitで既存の手法に比べて良好な性能を示していますが、本ブログ記事では割愛します。
Table1: structure-hard設定でのGenSPARCのContact予測ベンチマーク結果抜粋
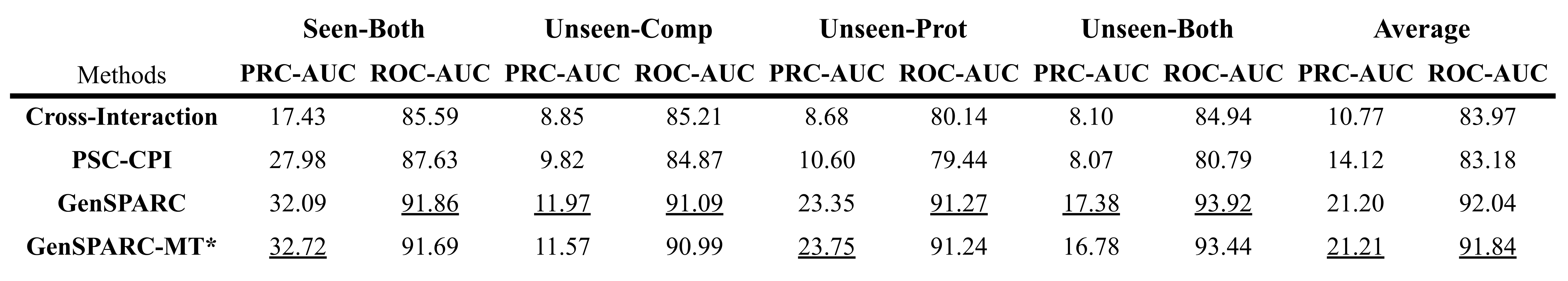
Table2: structure-hard設定でのGenSPARCの結合親和性予測ベンチマーク結果抜粋 (RMSE = root mean square error, PCCs = Pearson’s correlation coefficient)
バーチャルスクリーニング (DUD-E) での評価
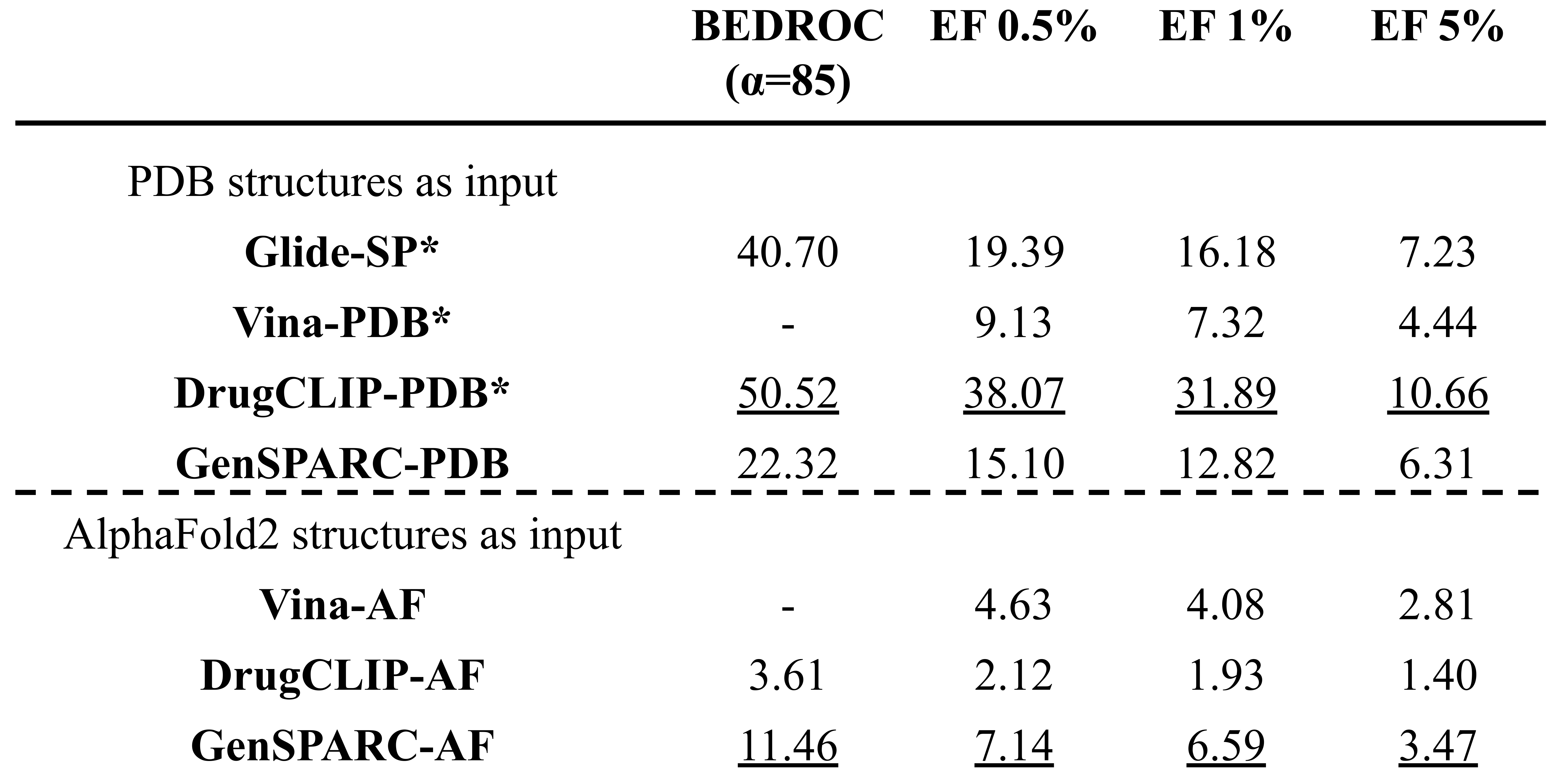
構造ベース仮想スクリーニングの代表的ベンチマークDUD-Eでも評価を実施しました。AutodockVina、DrugCLIP[12]、GenSPARCについては推論時の入力構造を、実験構造 (PDB) とAlphaFold2予測構造 (AF2) の2条件で比較しています。指標としてはEnrichment Factor (EF) とBoltzmann-enhanced discrimination of receiver operating characteristic (BEDROC) 指標を利用しています。
- PDB 構造を入力した場合: DrugCLIP にはわずかに及ばないものの、EF/BEDROC ともに競合レベル
- AlphaFold2 の予測構造を入力した場合: docking (Vina-AF) やDrugCLIP-AFより高いEF/BEDROCを達成
特に、PDB構造を入力した場合とAF2予測構造を入力した場合の性能ギャップが比較的小さいことが確認されました。これは、GenSPARCが3Diアルファベットという粗視化された構造表現を用いているため、原子座標の誤差に対して頑健であることを示唆しています。
Table 3: DUD-Eを用いたバーチャルスクリーニングのベンチマーク結果抜粋 (*付きの条件はDrugCLIP[12]の論文より数値を引用)
構造と物性を取り込む効果検証 (Ablation study)
GenSPARCの各要素の重要性を確認するため、構造認識SaProt、GCN、物性エンコーダのそれぞれを取り除いたAblation Studyを実施しました (Fig. 4)。
- 構造情報を抜く(=ESM-2に置き換え): 特にsequence-hard / structure-hard設定のUnseen-BothでコンタクトPRC-AUCが大きく悪化。
- GCNを抜く(=SPMMだけを用いる): ROC-AUCの減少幅は限定的だが、PRC-AUCが大きく低下。これは偽陽性が増加したことを示唆。
- 物性エンコーダを抜く(=GCNのみを用いる): 減少幅は小さいが、難しいセッティングでやや悪化。
この結果は、(1) タンパク質の配列+構造、(2) 化合物のグラフ構造と物性値、(3) それらをCross-Attentionで密につなぐという全要素の統合が、高い汎化性能の発現に不可欠であることを示しています。
 Fig. 4 GenSPARC各要素のAblation studyの結果
Fig. 4 GenSPARC各要素のAblation studyの結果
応用例や課題と今後の展望
GenSPARCは、実務的な創薬パイプラインにおいて以下の状況で有用性を発揮します。
- AlphaFold予測構造のみのターゲットに対する仮想スクリーニング:既存ドッキング手法と比較して予測構造に対する性能低下が小さく、安定したスコアリングが可能
- 大規模ライブラリの事前フィルタリング:GPUによる高速なバッチ計算を活用し、数百万〜数十億スケールのライブラリから有望な候補を絞り込み、ドッキングやMDシミュレーションの計算資源を最適化
- 構造ベース設計との統合:GenSPARCが算出したコンタクトマップを「有効性の高い領域」としてガイド情報に利用し、ポケット周辺のアミノ酸変異やリガンド設計にフィードバック
一方で、論文内ではいくつかの課題も議論しています。
- 化合物表現の限界:SPMMの物性値特徴量はRDKitベースの記述子が中心であり、3次元構造の微細な差異を十分に捉えきれていません。3次元情報をより多く取り入れたい一方で、座標を直接利用すると予測構造のノイズに対して過学習しやすいというトレードオフが存在します。
- コンタクトの予測漏れ:一部の相互作用については、既存手法より改善は見られるものの、依然として正確な予測ができていません。今後は、GenSPARCの予測コンタクトをAlphaFold3などの複合体予測モデルへの制約として適用し、構造予測とスコアリングを反復的に行う統合フレームワークを検討します。
- 天然変性タンパク質(IDP)への対応:現在のデータセットにはIDPがほとんど含まれていないため、これらの標的への適用性の検証と拡張が今後の重要な研究課題となります。
おわりに
AlphaFold2以降、「構造をどう使うか?」は創薬AIにとって中心的なテーマとなっています。GenSPARCは、構造認識タンパク質言語モデルSaProtと、化合物言語モデルSPMM+GCNを組み合わせ、配列・構造・物性を統合した次世代のCPI予測モデルです。複数のベンチマークで高い汎化性能を示し、特に「未知の相互作用ペア」や「AlphaFold予測構造を用いた仮想スクリーニング」でその強みを発揮します。今後は、化合物側の3D表現の改善や複合体構造予測モデルとの統合、IDPへの拡張などを通じて、「構造情報をフル活用しつつ、予測構造のノイズにも頑健」な次世代CPI予測へと発展させていくことが期待されます。
なお、本研究は2024年度のPFN夏季インターンシッププログラム期間中に行われた成果です。今回の論文の主著者のYiming Zhangさんからも、2024年PFN夏季インターンについて下記のコメントをいただきました。
PFNの強力な計算資源と、メンターの富田さん、武本さんの手厚いサポートのおかげで、短期間で研究を形にすることができました。自由な雰囲気の中で、社員の方々とランチや議論を楽しめたことも良い思い出です。このインターンで得た経験を、今後の研究活動に活かしていきたいと思います。
参考文献
1. Karimi M, Wu D, Wang Z, Shen Y. DeepAffinity: interpretable deep learning of compound-protein affinity through unified recurrent and convolutional neural networks. Bioinformatics. 2019;35: 3329–3338.
2. Nguyen T, Le H, Quinn TP, Nguyen T, Le TD, Venkatesh S. GraphDTA: predicting drug-target binding affinity with graph neural networks. Bioinformatics. 2021;37: 1140–1147.
3. Chen L, Tan X, Wang D, Zhong F, Liu X, Yang T, et al. TransformerCPI: improving compound-protein interaction prediction by sequence-based deep learning with self-attention mechanism and label reversal experiments. Bioinformatics. 2020;36: 4406–4414.
4. Lin Z, Akin H, Rao R, Hie B, Zhu Z, Lu W, et al. Evolutionary-scale prediction of atomic-level protein structure with a language model. Science. 2023;379: 1123–1130.
5. Notin P. Have We Hit the Scaling Wall for Protein Language Models? In: Pascal Notin [Internet]. 8 May 2025 [cited 9 Jun 2025]. Available: https://pascalnotin.substack.com/p/have-we-hit-the-scaling-wall-for
6. Su J, Han C, Zhou Y, Shan J, Zhou X, Yuan F. SaProt: Protein language modeling with structure-aware vocabulary. bioRxiv. 2023. p. 2023.10.01.560349. doi:10.1101/2023.10.01.560349
7. van Kempen M, Kim SS, Tumescheit C, Mirdita M, Lee J, Gilchrist CLM, et al. Fast and accurate protein structure search with Foldseek. Nat Biotechnol. 2024;42: 243–246.
8. Chang J, Ye JC. Bidirectional generation of structure and properties through a single molecular foundation model. Nat Commun. 2024;15: 2323.
9. Karimi M, Wu D, Wang Z, Shen Y. Explainable deep relational networks for predicting compound-protein affinities and contacts. J Chem Inf Model. 2021;61: 46–66.
10. You Y, Shen Y. Cross-modality and self-supervised protein embedding for compound-protein affinity and contact prediction. Bioinformatics. 2022;38: ii68–ii74.
11. Wu L, Huang Y, Tan C, Gao Z, Hu B, Lin H, et al. PSC-CPI: Multi-scale Protein Sequence-structure contrasting for efficient and generalizable compound-Protein Interaction prediction. arXiv [q-bio.BM]. 2024. Available: http://arxiv.org/abs/2402.08198
12. Gao B, Qiang B, Tan H, Ren M, Jia Y, Lu M, et al. DrugCLIP: Contrastive protein-molecule representation learning for virtual screening. arXiv [cs.LG]. 2023. Available: http://arxiv.org/abs/2310.06367