Blog
Optuna開発メンバの太田(@sile)です。この記事では、Optuna開発時に利用している拙作のkurobakoというツールの紹介をさせていただきます。
kurobakoを一言で表すなら「Optunaの(ほぼ)全機能に対応した、ブラックボックス最適化向けベンチマークツール」となります。
Optunaとブラックボックス最適化
Optuna [1]は、PFNが開発しているオープンソースのハイパーパラメータ自動最適化フレームワークです。Optunaにはsamplerおよびprunerと呼ばれる構成要素が存在し、これらを変更することで、最適化アルゴリズムをカスタマイズすることが可能になっています。
samplerは、ユーザが定義した目的関数の結果の最小化または最大化を目指し、次のトライアルで評価するハイパーパラメータを決定します(図1)。その際には、対象の目的関数に特別な性質(例えば微分可能性)は要求せずに、過去のハイパーパラメータの評価結果だけを考慮して動作します。このような最適化アルゴリズムは、ブラックボックス最適化アルゴリズムとも呼称されます。
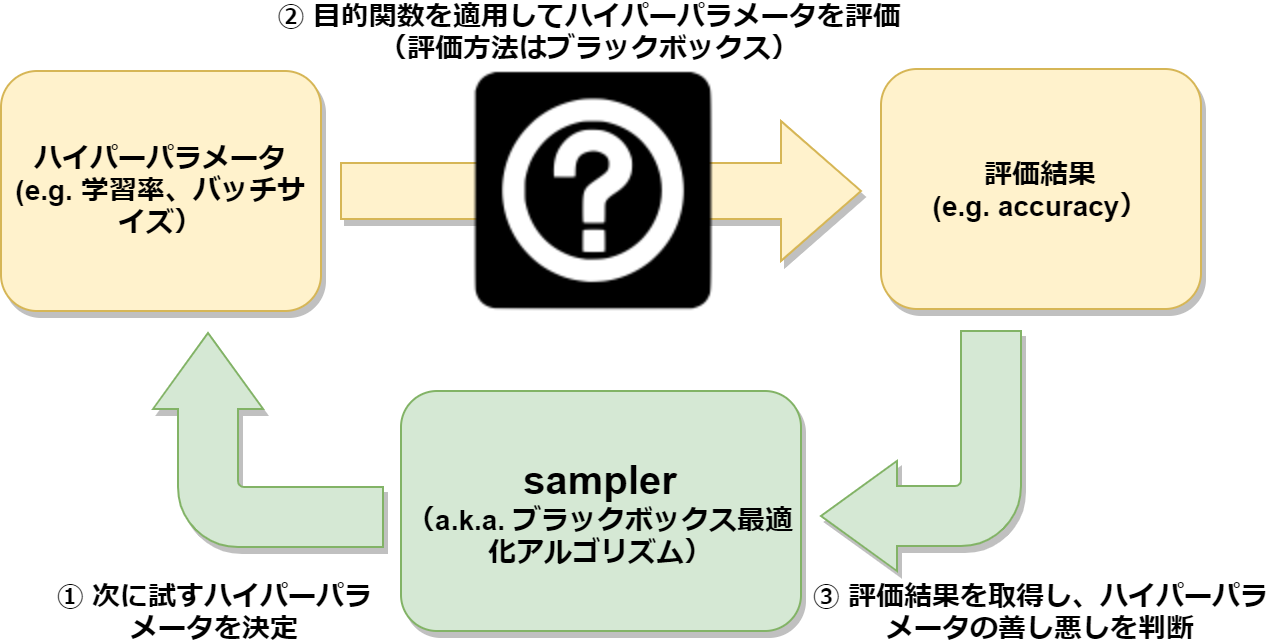
図1 samplerとブラックボックス最適化
一方で、prunerはハイパーパラメータの評価を途中で打ち切るかどうかを決定します。例えば、深層学習でモデルを訓練する際のハイパーパラメータを最適化したいとします。この場合、ハイパーパラメータの一回の評価(モデルの訓練)に長時間を要することは珍しくありません。そこでprunerを用いて、明らかに性能が悪いモデルは完全には評価(訓練)せずに、初期段階で中止するようにすれば、最適化に掛かる時間を短縮することが可能になります。なお、ブラックボックス最適化の文脈では、samplerとprunerといった明確な区別は設けずに、両者を合わせて、multi-fidelityブラックボックス最適化といった枠組みで扱われることが多いです(図2)。
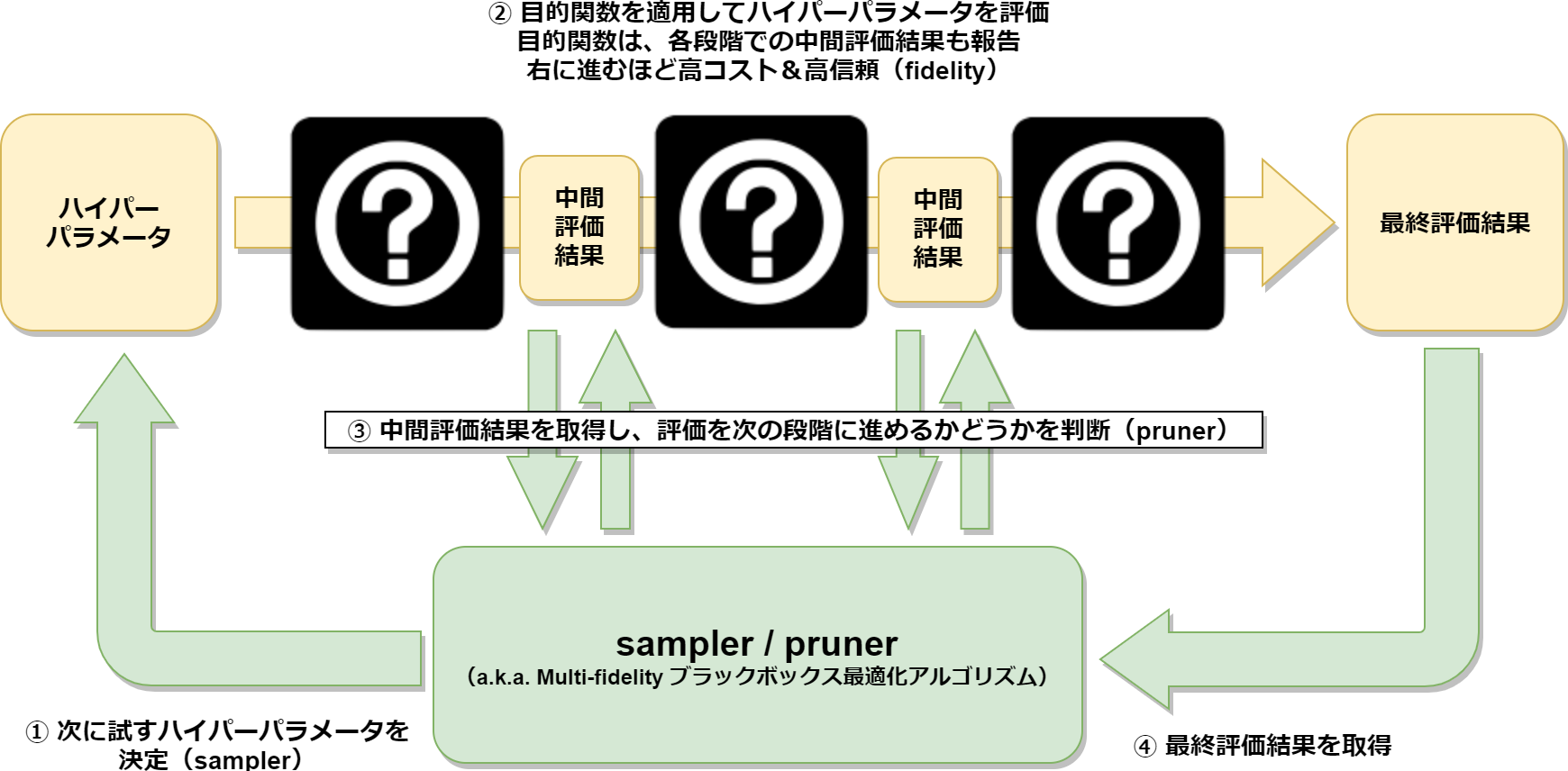
図2 sampler/prunerとmulti-fidelityブラックボックス最適化
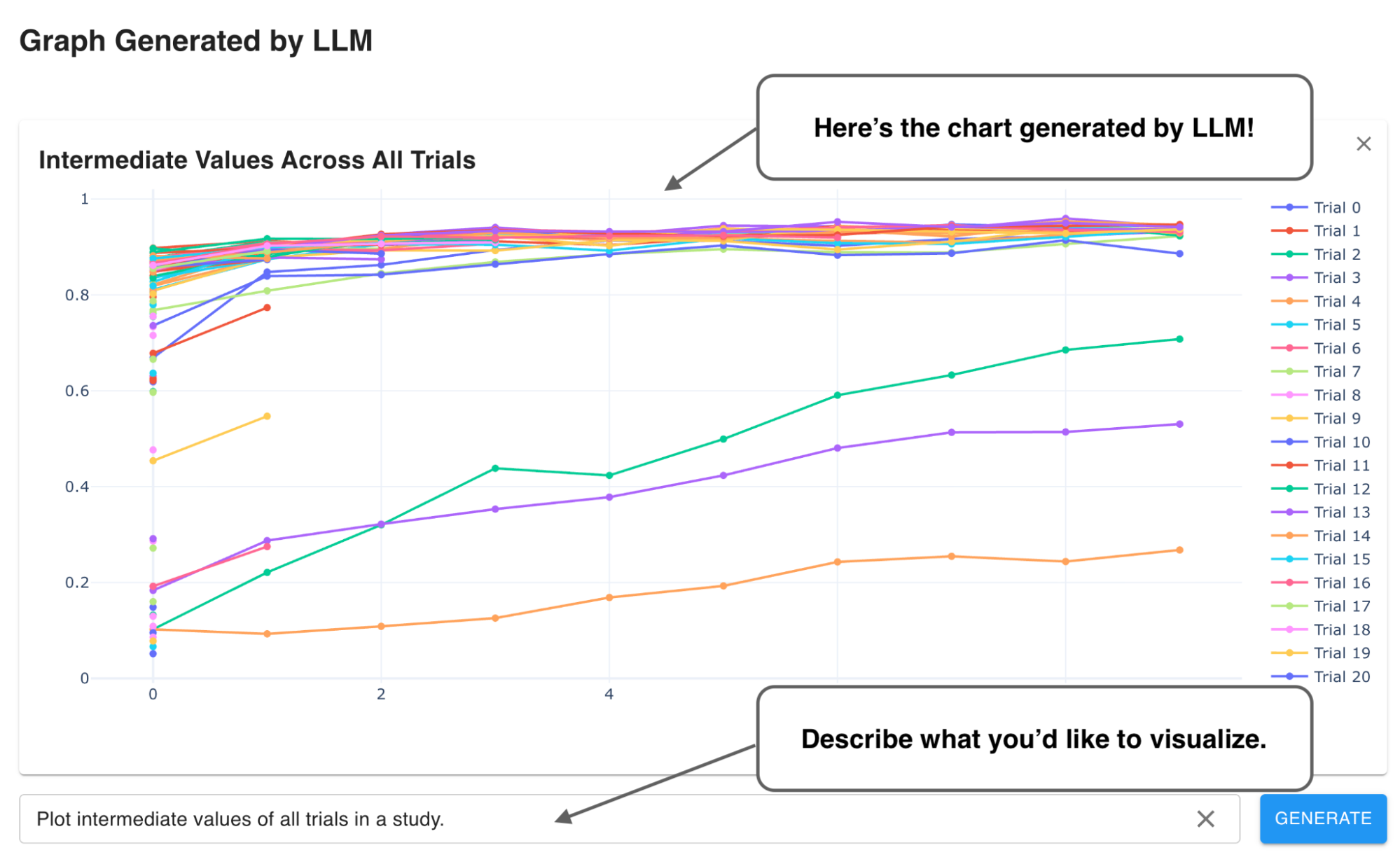
kurobakoは、このmulti-fidelityブラックボックス最適化に対応したベンチマークツールとなっています。
開発の動機
Optunaの開発に携わっていると、samplerやprunerのアルゴリズムの改善や試作を行う機会がたびたびあります。その際に難しいのが「新しいアルゴリズムの最適化性能の評価をどう行うか」といった問題です。
ブラックボックス最適化自体は歴史のある研究分野なので、最適化アルゴリズムの性能評価に使える様々なベンチマーク関数 [2]が開発されています。また、そのようなベンチマーク関数を利用したものとして、例えばCOCO (COmparing Continuous Optimisers) [3]というOSSのベンチマークフレームワークも存在します(図3)。ただし、これらの関数の多くは「連続変数で構成された探索空間」かつ「単一fidelity」のみを扱っており、Optunaが想定している問題設定のサブセットしかカバーされていません。
図3 COCOに含まれるベンチマーク関数の一部(一覧)
より現実的な問題を扱ったベンチマークツールとしては、今年にGitHubで公開されたnas_benchmarksがあります。これはニューラルネットワークのアーキテクチャ探索(NASBench)[4]およびハイパーパラメータ探索(HPOBench)[5]という二種類のタスクに基づくベンチマークを提供しており、その探索空間は離散変数とカテゴリ変数から構成されています(図4)。加えて、multi-fidelityな評価にも対応しています。
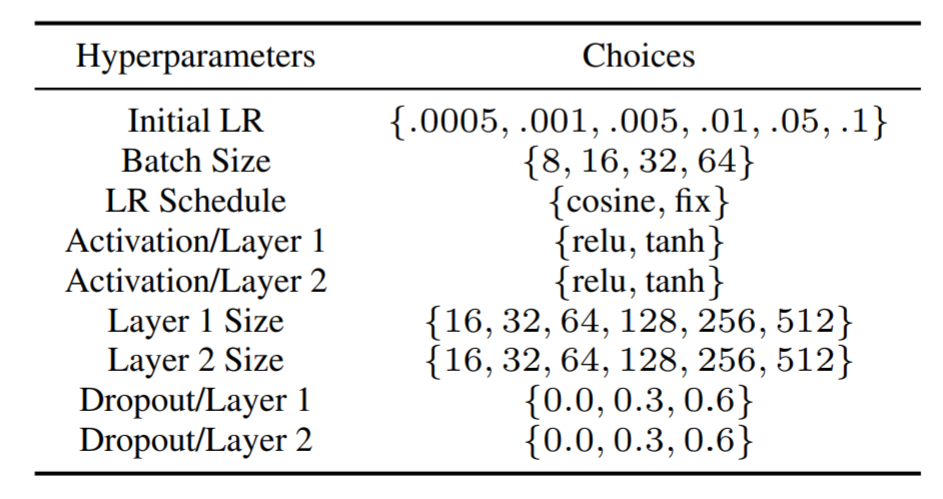
図4 HPOBenchの探索空間 [5]
上述のベンチマーク群は、それぞれ有用なものではありますが、これだけで事足りるかといえば、そうではありません。
具体的には、Optunaに含まれている最適化アルゴリズム関連の主要機能を一通り評価するためには、以下がサポートされている必要があります(括弧内は対応するOptunaの機能名):
- 連続・離散・カテゴリ変数を含んだ探索空間(suggest_{uniform, int, categorical})
- multi-fidelityな評価(枝刈り)
- 複数トライアルの非同期並行実行(分散最適化)
- ifやforループを含んだ探索空間(define-by-run)
私の知る限りでは、上記の全てに対応したベンチマークツールは存在しませんでした。これらをカバーし、Optunaの全機能を簡単に評価できるようにしたかった、というのがkurobakoの開発動機です。
また、特定のベンチマークに絞って性能評価を行い続けると、最適化アルゴリズム(およびそれ自体のハイパーパラメータ)が、そのベンチマークにオーバーフィットしてしまう危険性があります。それを防ぐために、新しいベンチマーク問題の追加が簡単で、多様なベンチマーク群を同じ枠組みで扱えるようにしておきたかった、というのもモチベーションの一つです。
現時点では、NASBenchとHPOBenchに加えて、sigopt/evalset [6]で提供されているベンチマーク関数が、kurobakoに標準で組み込まれています。
オープンな実例としてはMake TPESampler pruning aware #439やAdd CMA-ES Sampler #447といったPRで、最適化性能評価にkurobakoが使用されています。
基本的な使用方法
kurobakoは、Rustで書かれたコマンドラインツールで、入出力フォーマットにはJSONを採用しています。
以下に、基本的なベンチマークの実行例を掲載します:
# (1) kurobakoのバイナリをダウンロード
$ curl -L https://github.com/sile/kurobako/releases/download/0.1.2/kurobako-0.1.2.linux-amd64 -o kurobako
$ chmod +x kurobako && sudo mv kurobako /usr/local/bin/
# (2) HPOBench用のデータファイルをダウンロード ※ サイズは700MB程度
$ curl -OL http://ml4aad.org/wp-content/uploads/2019/01/fcnet_tabular_benchmarks.tar.gz
$ tar xf fcnet_tabular_benchmarks.tar.gz && cd fcnet_tabular_benchmarks/
# (3) 使用するベンチマーク問題を指定
#
# 今回は、HPOBenchの中から
# "protein structure"および"parkinsons telemonitoring"
# というデータセットを用いた二つのベンチマークを採用
$ kurobako problem hpobench fcnet_protein_structure_data.hdf5 > problems.json
$ kurobako problem hpobench fcnet_parkinsons_telemonitoring_data.hdf5 >> problems.json
$ cat problems.json
{"hpobench":{"dataset":"fcnet_protein_structure_data.hdf5"}}
{"hpobench":{"dataset":"fcnet_parkinsons_telemonitoring_data.hdf5"}}
# (4) 使用する最適化アルゴリズム(kurobako用語では”solver”)を指定
#
# 今回は、Optunaが提供しているアルゴリズムの中から以下の四つを採用:
# - RandomSampler(枝刈りなし)
# - TPESampler(枝刈りなし)
# - TPESampler + MedianPruner
# - TPESampler + SuccessiveHalvingPruner
$ kurobako solver --name 'RandomSampler' optuna --sampler random --pruner nop > solvers.json
$ kurobako solver --name 'TPESampler' optuna --sampler tpe --pruner nop >> solvers.json
$ kurobako solver --name 'TPESampler_with_MedianPruner' optuna --pruner median --median-warmup-steps 4 >> solvers.json
$ kurobako solver --name 'TPESampler_with_SuccessiveHalvingPruner' optuna --pruner asha --asha-min-resource 4 >> solvers.json
$ cat solvers.json
{"name":"RandomSampler","optuna":{"sampler":"random","pruner":"nop"}}
{"name":"TPESampler","optuna":{"pruner":"nop"}}
{"name":"TPESampler with MedianPruner","optuna":{"median_warmup_steps":4}}
{"name":"TPESampler with SuccessiveHalvingPruner","optuna":{"pruner":"asha","asha_min_resource":4}}
# (5) ベンチマークを実行
#
# 乱数によるブレを抑えるために、同じsolver/problemの組のベンチマーク実行を三十回繰り返す
$ kurobako studies --solvers $(cat solvers.json) --problems $(cat problems.json) --repeats 30 --budget 80 > studies.json
$ head -1 studies.json
{"solver":{"name":"RandomSampler","optuna":{"sampler":"random","pruner":"nop"}},"problem":{"hpobench":{"dataset":"fcnet_protein_structure_data.hdf5"}},"budget":80,"concurrency":1,"scheduling":"RANDOM"}
$ cat studies.json | kurobako run --parallelism 10 > result.json
(ALL) [01:27:14] [STUDIES 240/240 100%] [ETA 0s] done
# (6) ベンチマーク結果レポートを生成
$ cat result.json | kurobako report
...長いので省略...
最後のレポートの内容はこのgistに貼ってあります。
ベンチマーク結果(result.json)から図を生成することも可能です:
$ cat result.json | kurobako plot curve
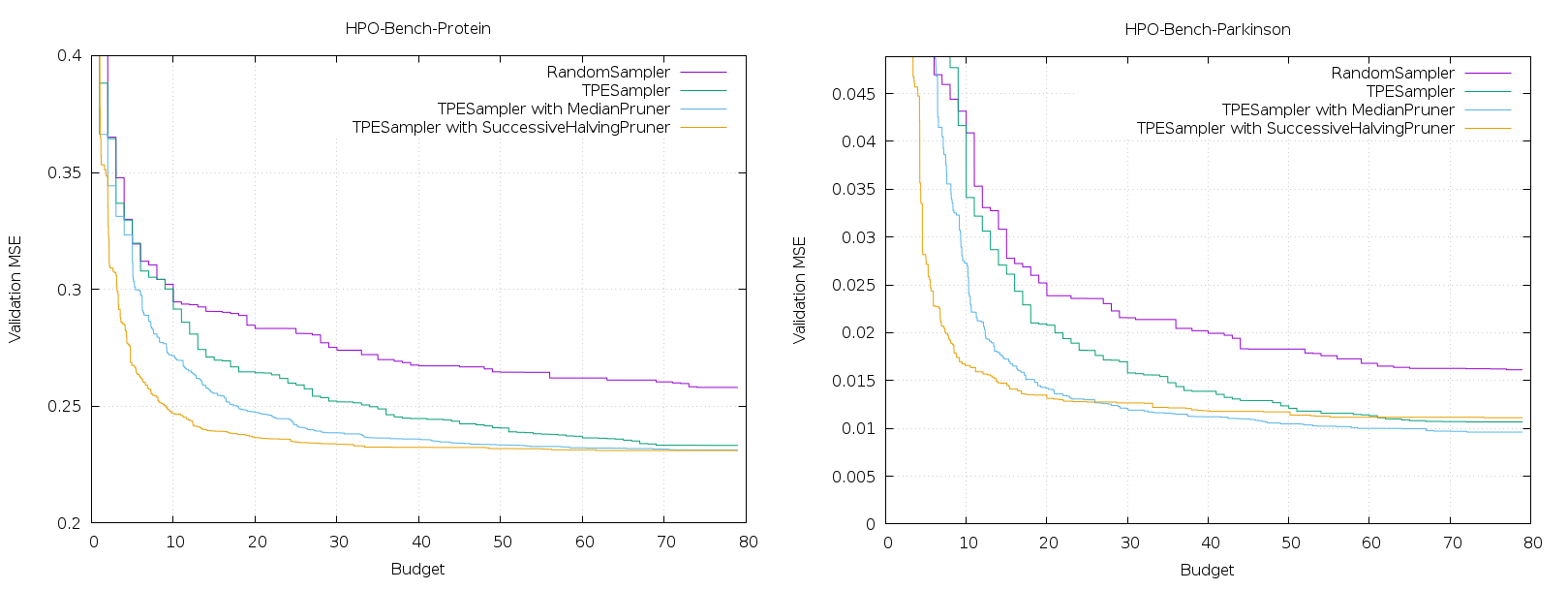
図5 ベンチマーク結果
図のY軸は目的関数のベスト値(三十回平均)です。X軸は最適化時のリソース消費量を表し、一バジェットが「目的関数の一回の完全な評価」に対応します。HPOBenchでは、完全な評価(モデルの訓練)に100エポック掛かるので、例えばバジェットの値が40なら、4000エポック分の訓練を実行していることになります。なおHPOBenchでは、事前に探索空間内の全てのハイパーパラメータの組み合わせを評価して、その結果がキャッシュされているので、ベンチマーク実行時には実際のモデルの訓練は不要で、キャッシュ参照だけで済むようになっています。
上のベンチマークは一例に過ぎませんが、利用可能なバジェットが少ない場合には、枝刈りを有効にした方が良好な結果が得られていることが見て取れるかと思います。ただし、利用可能なバジェットが増えるにつれて枝刈りの効果は弱まり、特に”parkinsons telemonitoring”データセットの方では終盤で、SuccessiveHalvingアルゴリズムによる枝刈りありのTPE [7]が、枝刈りなしのものに追い抜かされています。
最適化アルゴリズムの追加方法
kurobako自体はRust製ですが、独自の最適化アルゴリズム(solver)を、コマンドラインプログラムとして実装することが可能になっています(problemも同様)。
Python向けには、PyPIに同じ名前のkurobakoというパッケージが用意されているので、それを使えば以下のようにしてsolverが実装できます:
# filename: random.py
#
# ランダム探索に基づくsolver実装
from kurobako import problem
from kurobako import solver
import numpy as np
class RandomSolverFactory(solver.SolverFactory):
def specification(self):
return solver.SolverSpec(name='Random Search')
def create_solver(self, seed, problem):
return RandomSolver(seed, problem)
class RandomSolver(solver.Solver):
def __init__(self, seed, problem):
self._rng = np.random.RandomState(seed)
self._problem = problem
# 次に試すハイパーパラメータを問い合わせる
def ask(self, idg):
trial_id = idg.generate()
next_step = self._problem.steps.last_step
params = []
for p in self._problem.params:
if p.distribution == problem.Distribution.LOG_UNIFORM:
low = np.log(p.range.low)
high = np.log(p.range.high)
params.append(float(np.exp(self._rng.uniform(log, high))))
else:
params.append(self._rng.uniform(p.range.low, p.range.high))
return solver.NextTrial(trial_id, params, next_step)
# ハイパーパラメータの評価結果を通知する(ランダム探索では関係なし)
def tell(self, trial):
pass
if __name__ == '__main__':
runner = solver.SolverRunner(RandomSolverFactory())
runner.run()
対象の最適化アルゴリズムが、Optunaのsamplerインターフェースに準拠している場合は、より簡単です:
# filename: sa.py
#
# SimulatedAnnealingに基づくsolver実装
from kurobako import solver
from kurobako.solver.optuna import OptunaSolverFactory
import optuna
class SimulatedAnnealingSampler(optuna.BaseSampler):
# 実装は https://github.com/optuna/optuna/blob/v0.19.0/examples/samplers/simulated_annealing_sampler.py を参照
...
def create_study(seed):
sampler = SimulatedAnnealingSampler(seed=seed)
return optuna.create_study(sampler=sampler)
if __name__ == ‘__main__’:
factory = OptunaSolverFactory(create_study)
runner = solver.SolverRunner(factory)
runner.run()
これらのsolverは、以下の様にして使用できます:
$ kurobako solver command python random.py > solvers.json $ kurobako solver command python sa.py >> solvers.json $ kurobako studies --problems $(cat problems.json) --solvers $(cat solvers.json) | kurobako run > result.json
おわりに
この記事ではkurobakoという、ブラックボックス最適化用のベンチマークツールを紹介させていただきました。「Optunaのアルゴリズム周りにコントリビュートしてみたいけれど、最適化性能がちゃんと出ているかが心配」といった人がいれば、是非試してみてください。最終的には、OptunaのCIの一部にベンチマークを組み込むところまで持っていけると嬉しいです。
この記事内では触れられませんでしたが、実はkurobakoは、現在のOptunaにはない枠組み(e.g., multi-objective、制約付き最適化、トライアルのsuspend/resume)のベンチマークにも対応しています。また少し変わった使い方をすれば「solver自体のハイパーパラメータ最適化(solverのproblem化)」や、さらに進んで「複数のproblemに跨って満遍なく良く動作するデフォルトハイパーパラメータの探索」といったメタ最適化的な用途で使用することも可能となっています。
まだドキュメント作成が追いついてないですが、順次追加予定なので、もし興味を持った方がいればGitHubのREADMEやWikiも参照していただけると幸いです。
参考文献
[1] Akiba, Takuya, et al. “Optuna: A next-generation hyperparameter optimization framework.” Proceedings of the 25th ACM SIGKDD International Conference on Knowledge Discovery & Data Mining. ACM, 2019.
[2] Jamil, Momin, and Xin-She Yang. “A literature survey of benchmark functions for global optimization problems.” arXiv preprint arXiv:1308.4008 (2013).
[3] Hansen, Nikolaus, et al. “COCO: A platform for comparing continuous optimizers in a black-box setting.” arXiv preprint arXiv:1603.08785 (2016).
[4] Ying, Chris, et al. “Nas-bench-101: Towards reproducible neural architecture search.” arXiv preprint arXiv:1902.09635 (2019).
[5] Klein, Aaron, and Frank Hutter. “Tabular Benchmarks for Joint Architecture and Hyperparameter Optimization.” arXiv preprint arXiv:1905.04970 (2019).
[6] Dewancker, Ian, et al. “A strategy for ranking optimization methods using multiple criteria.” Workshop on Automatic Machine Learning. 2016.
[7] Bergstra, James S., et al. “Algorithms for hyper-parameter optimization.” Advances in neural information processing systems. 2011.