Blog
本記事は、2024年夏季インターンシッププログラムで勤務された大村拓登さんによる寄稿です。
はじめに
2024年夏季インターンシップに参加した、東京大学大学院農学生命科学研究科修士課程2年の大村拓登です。大学では、量子化学計算を用いた酵素触媒の反応機構の解析や、酵素に特化した機械学習ポテンシャルモデルの開発といった研究に取り組んでいます。
今回のインターンシップでは「LightPFPの酵素反応への適用」というテーマに取り組みました。本インターンシップで取り組んだのは、主に以下の2点です。
- 酵素反応をPFPで扱う方法論の開発
- Matlantisの新機能であるLightPFPを酵素反応に適用できるかどうかの検証
背景
Matlantisの中核をなすPFPは、計算に多大な時間を要する量子力学に基づく原子レベルのシミュレーション結果を学習することで、高速なシミュレーションを可能にした「機械学習ポテンシャル」モデルです。PFPは、これまで主に結晶・金属・ポリマーなどの材料分野で利用されてきましたが、タンパク質のような生体高分子への適用例はほとんどありませんでした。
タンパク質、特に酵素は、生体中の化学反応を触媒する生体触媒であり、生命現象のほぼすべてに関与する重要な研究対象です。酵素研究は、医療・創薬、食品科学、農業、環境科学など、幅広い分野において応用可能であり、酵素反応を扱える分子シミュレーションツールへの需要は非常に大きいと考えられます。特に、PFPのような高速シミュレーションツールでの取り扱いが期待されています。
しかしながら、PFPはタンパク質のような生体高分子には適用されてきませんでした。これにはいくつかの理由※1が考えられますが、最も大きな理由は「PFPでも、タンパク質の全原子を扱った大規模なシミュレーションには時間がかかる」と想定されるためです。
酵素タンパク質は、比較的小さいものでも数千の原子からなる巨大分子(図1)で、複合体を考慮すると系に含まれる原子数は数万を超えることが想定されます。PFPで扱える最大の原子数は数万原子ですので、大きなタンパク質では取り扱いが困難となります。
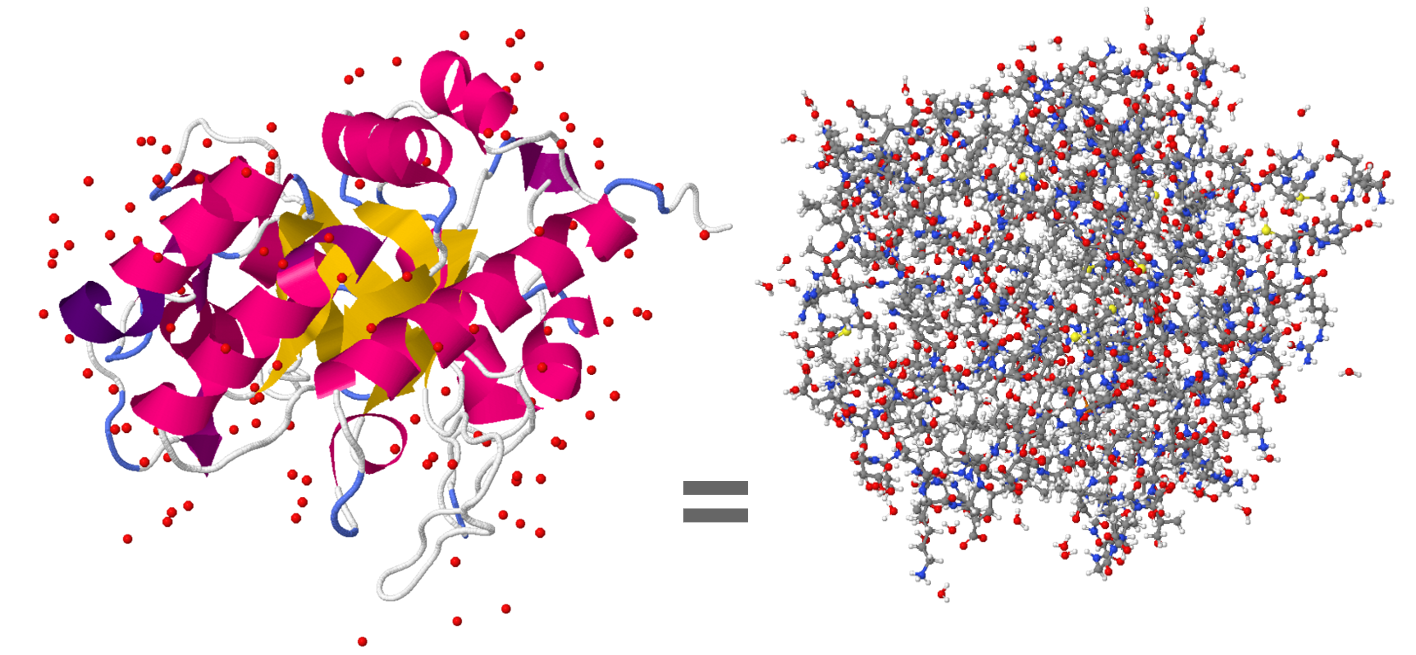
図1. 脱炭酸酵素(PDB ID: 1DQX [2])の3次元構造(1量体)。この酵素の場合は比較的小さい4561原子からなる。
生体高分子に限らず、このような巨大な系に対するPFPの限界を克服するために開発されたアプローチがLightPFPです。汎用モデルであるPFPに対して、LightPFPは任意の構造・系に特化させることで、計算コストを抑えつつ、高速(20~50倍)で、より大きいサイズ(~50万原子)の系を扱えるように設計されたモデルです。
基本的な使い方としては、ユーザーが扱いたい系の構造についてPFPで計算を行い、これを訓練データとしてLightPFPモデルをファインチューニングすることで、所望の系に特化したモデルを作成します。モデルの精度は訓練データに大きく依存するため、最も重要なのは、訓練データにどのような部分構造を用いるか、そしてどのようにデータを生成するかという点です。
LightPFPは本インターン直前に公開されたMatlantisの新機能であり、本インターン開始時点では、合金や固液界面、液ー液相分離のシミュレーションへの応用が検証されていました。応用分野はまだ開拓中であり、これまでPFPで扱われてこなかった分野、例えば酵素反応のような生体高分子を用いた系への応用が可能かどうかを検証することが、本インターンの目的となりました。
※1: 分子の大きさ以外の理由として、以下が考えられます。
・酵素反応のような巨大で複雑な系に対して、機械学習ポテンシャルモデルで反応障壁を求める方法論が確立されていない。
課題とアプローチ
今回の検証には大きな課題がありました。それは、そもそも酵素反応に関するPFPでの計算データが存在しないという点です。このため酵素に特化したLightPFPモデルを作成しても、参照データがなくその妥当性を検証することができませんでした。
そこで、まずはPFPを用いて酵素反応を扱えるかを検証することが必要となりました。幸い、数千原子からなる比較的小さな酵素であればPFPでも取り扱いが可能であるため、まずはPFPを用いて酵素反応を扱えるかを確認し、その計算結果を基に、LightPFPで再現できるかを検証するという流れで進めました。
手法
ターゲットとする酵素反応
本インターンではケーススタディの対象として、脱炭酸酵素(ODCase)を選択しました。この酵素は4,561原子(※1量体の場合)と比較的小さい上に、その反応は分子(OMP:Orotidine-5′-MonoPhospathase)からカルボキシ基を脱離させるという比較的シンプルなものです。酵素中ではOMP分子からカルボキシ基が脱離したのち、根元のカルボアニオンに対して、隣接した塩基性アミノ酸であるリシン(Lys 93)から水素原子が供与されます。
OMP分子の脱炭酸反応の自由エネルギー障壁ΔG‡の値について、先行研究[1]で以下のように実験値とQM/MMによるメタダイナミクスでの計算値が求められているので、これらをPFPによる計算の参照値としました。
- 酵素中:実験値 17 kcal/mol,計算値 33 kcal/mol
- 水溶液中:実験値 39 kcal/mol,計算値 40 kcal/mol
初期構造の準備
Matlantis上でこの酵素反応を扱うために、タンパク質ー基質分子複合体の構造を用意する必要がありました。まずはタンパク質の3次元構造のデータベースであるProtein Data Bank(PDB)に登録されている構造(PDBファイル)を編集して、1量体のタンパク質構造を作成しました[2]。ここで、生体中でのアミノ酸のイオン化状態を考慮して水素を付加しました。次にこのPDBファイルに含まれる基質分子を編集してOMP分子に変更し、水分子を酵素が取り囲むように配置しました。最後に、系全体の電荷が0になるようにイオンを配置しました。
また水溶液での反応と比較して酵素がどれだけ反応障壁を下げるかを検証するために、水溶液中での反応障壁の計算を行いました。ここではexplicit溶媒を利用し、水分子で満たした周期境界セル中にOMP分子を配置しました。また、系全体の電荷が0になるようにイオンを配置しています。なおLightPFPで部分構造を学習することを考慮すると、元素の種類が少ない方が都合がいいので、今回はカウンターイオンにH3O+を用いています。本来は比較的不活性なNa+を用いたほうが妥当です。
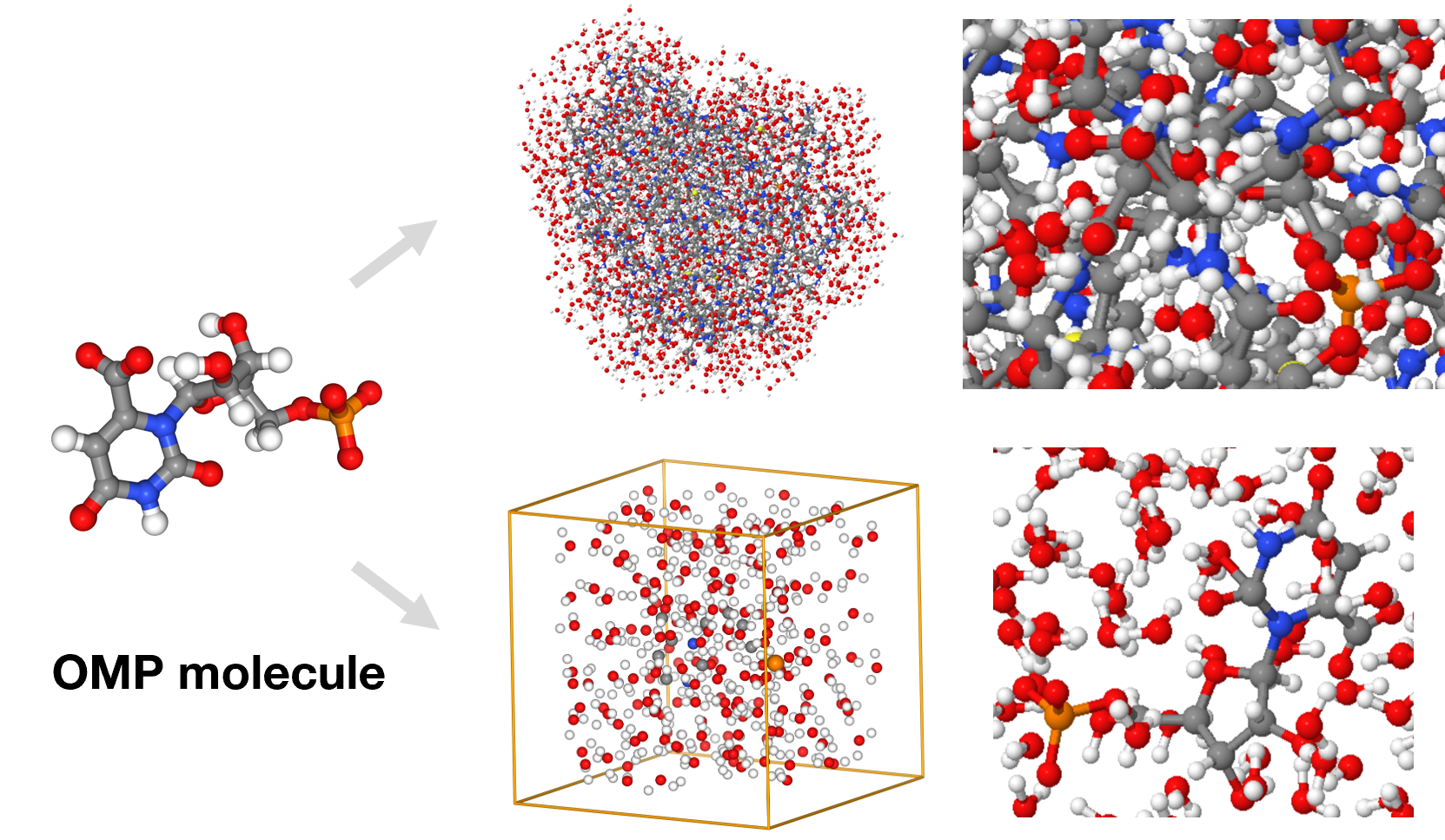
図2. 酵素中(上)と水溶液中(下)の初期構造。
反応障壁の計算
PFPおよびLightPFPの両方について、以下の手法で反応障壁を求めました。
反応障壁の計算のために、OMPとCO2の結合距離を少しずつ伸ばしていき、その都度結合距離だけ固定して構造最適化を行うScanを実施しました(図3上)。このScanで得られたトラジェクトリをMatlantisに実装されているReactionString法の入力として、反応経路を決定しました(図3下)。この反応経路のうち、脱炭酸反応に該当するピークについて、エネルギー障壁を求めました。
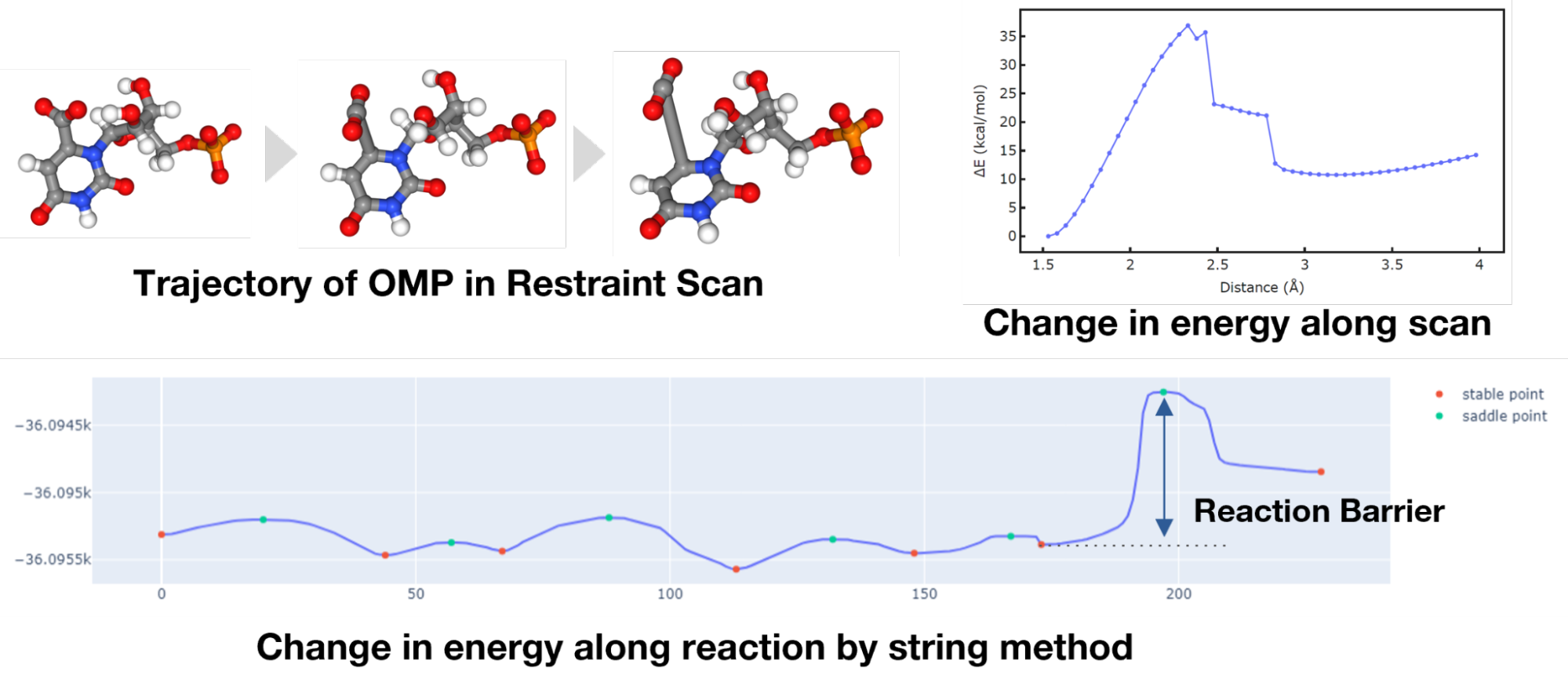
図3.反応障壁を求める際に用いたScanによる構造の変化(左上)と得られたエネルギーの遷移の例(右上)、およびReactionString法で計算した活性化エネルギー(直前のstable pointとsaddle pointとのエネルギー差)の例(下)。
LightPFPモデルの訓練
酵素とOMPから構成される系について、以下のようにLightPFPモデルのための学習データを用意しました。
全系を均等に学習対象として用いる場合、決められたカットオフ半径(5 Å, 10 Å, 15 Å)内の構造を全系からランダムに切り出し、それぞれに対して複数の温度(300 K, 500 K, 1000 K)で MDシミュレーションを実行し、サンプリングを行いました。
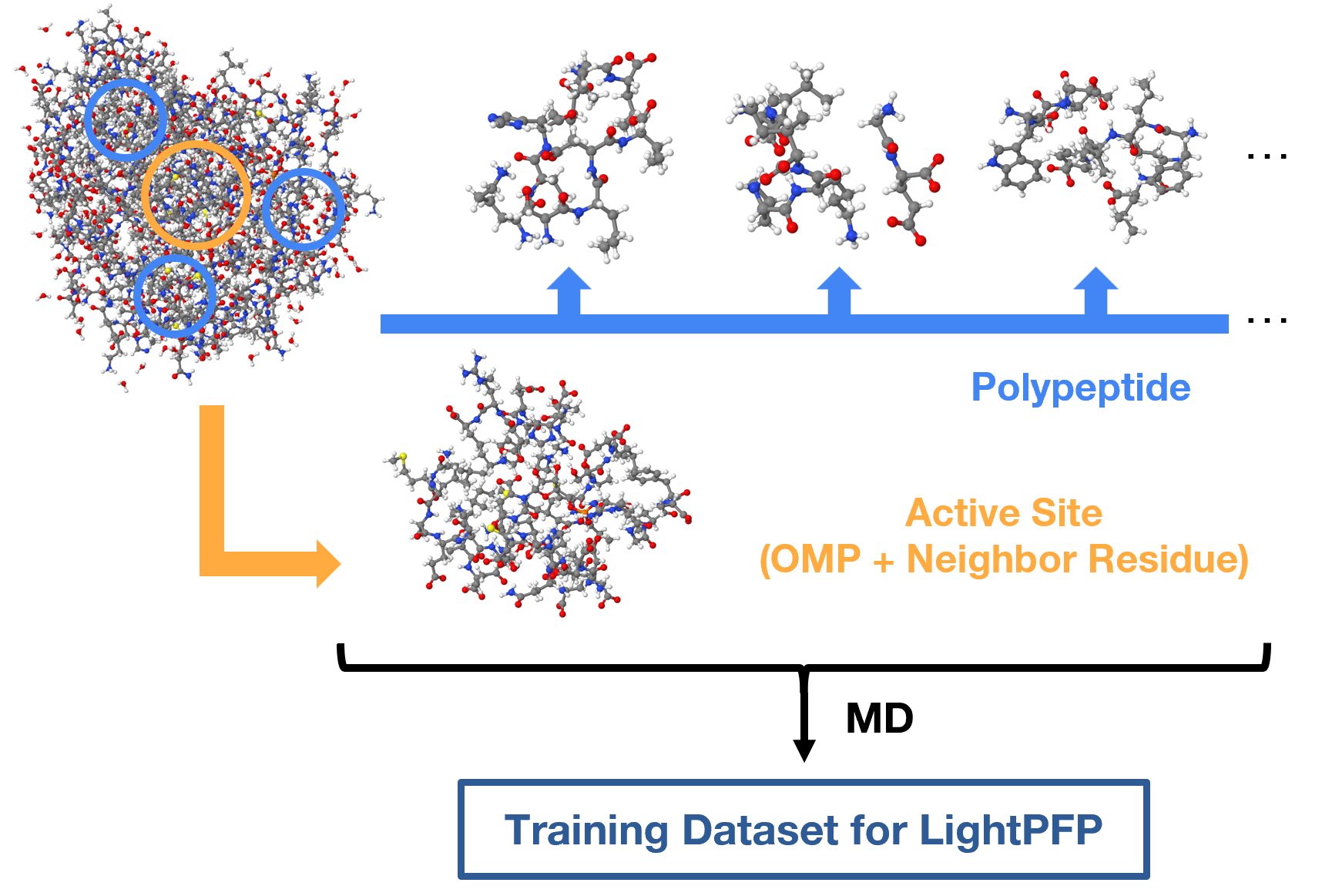
上記のランダムサンプリングとは別に、活性部位を明示的に選択して学習データとして用いる場合も検証しました。この際カットオフを5 Åとし、活性部位については複数の温度(300 K, 500 K, 1000 K)で MDシミュレーションを実行して各200点のサンプリングを行い、ポリペプチドについては300 KのMDシミュレーションで500点のサンプリングを行いました。
また学習にはLightPFPの事前学習済みモデル(ORGANIC_SMALL_NN)を用いました。
結果・考察
PFPによる反応障壁の計算
水溶液中の場合、以下のような反応経路が得られました。
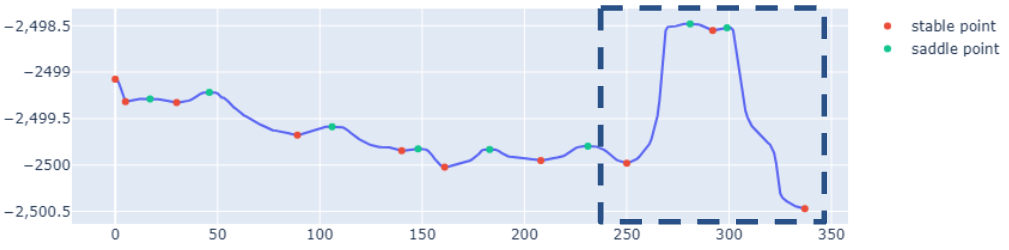
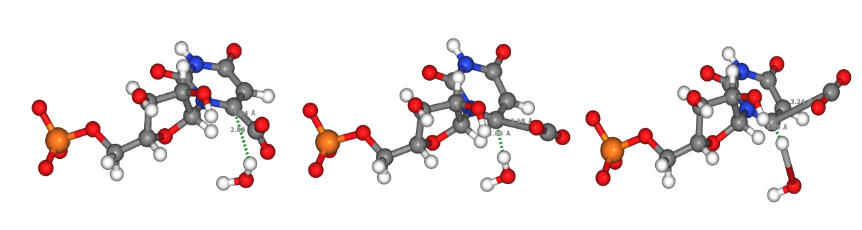
ここから酵素中での反応障壁を34.7 kcal/molと見積もることができました。参考文献[1]にある実験値が39 kcal/mol、DFTによる計算結果が40 kcal/molであり、先行研究よりも低い値となりました。また下図のように、水分子からの水素原子の供与が確認されました。
酵素中の場合、以下のような反応経路が得られました。
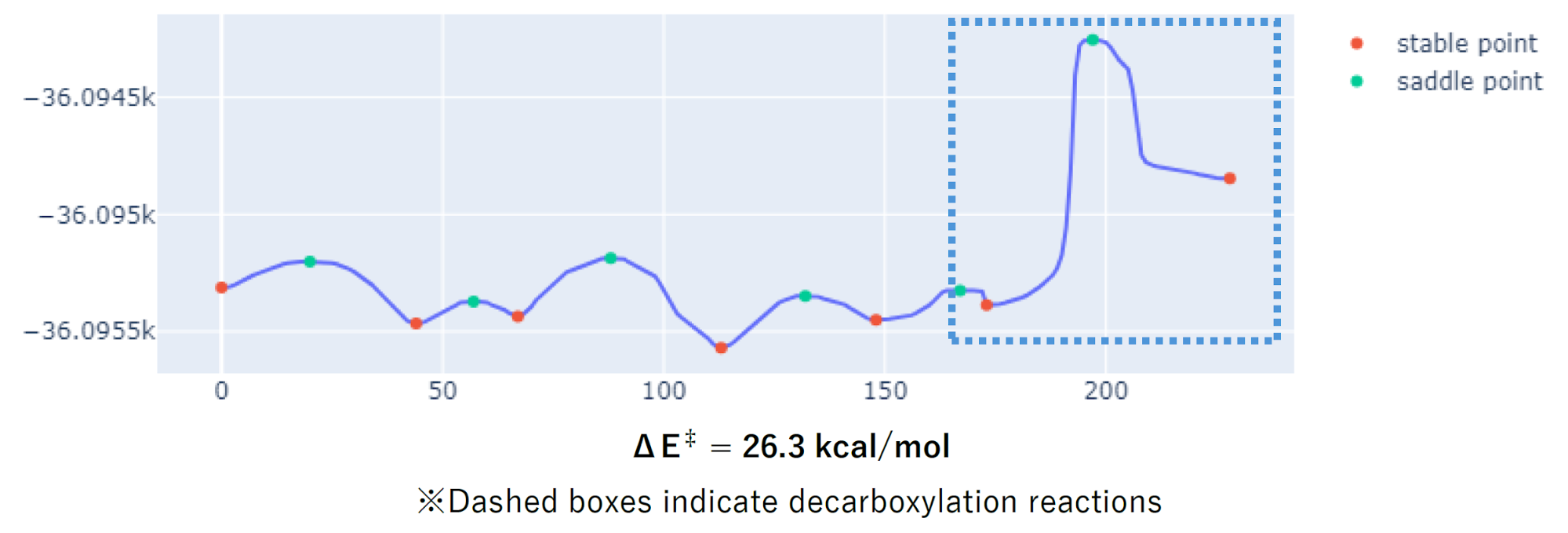
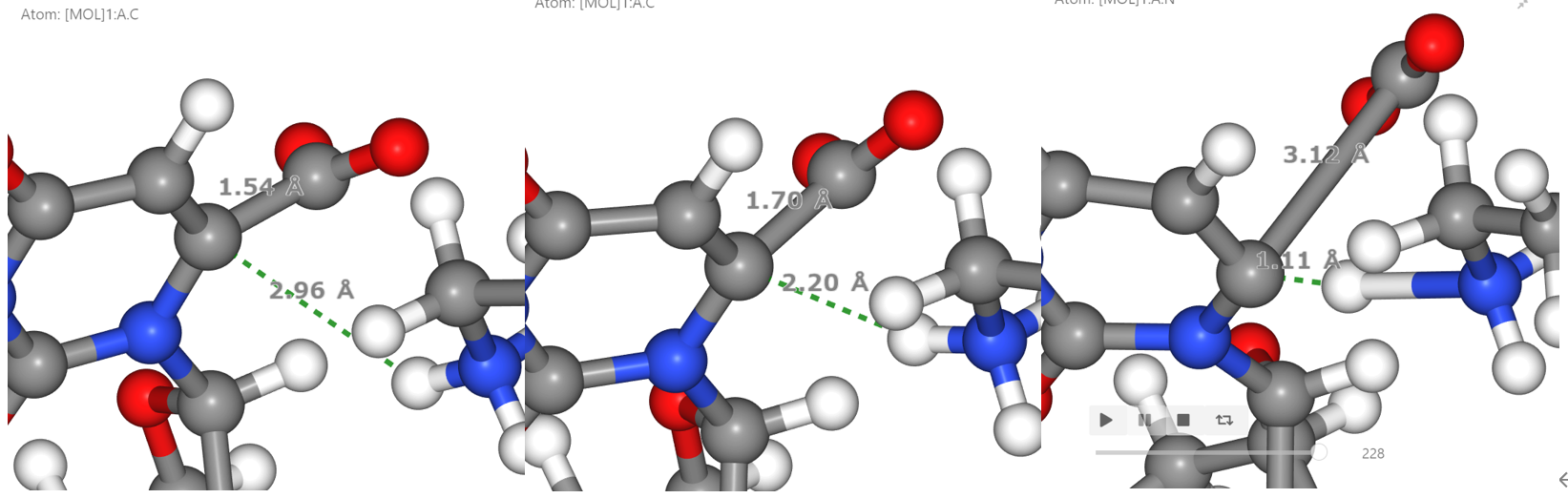
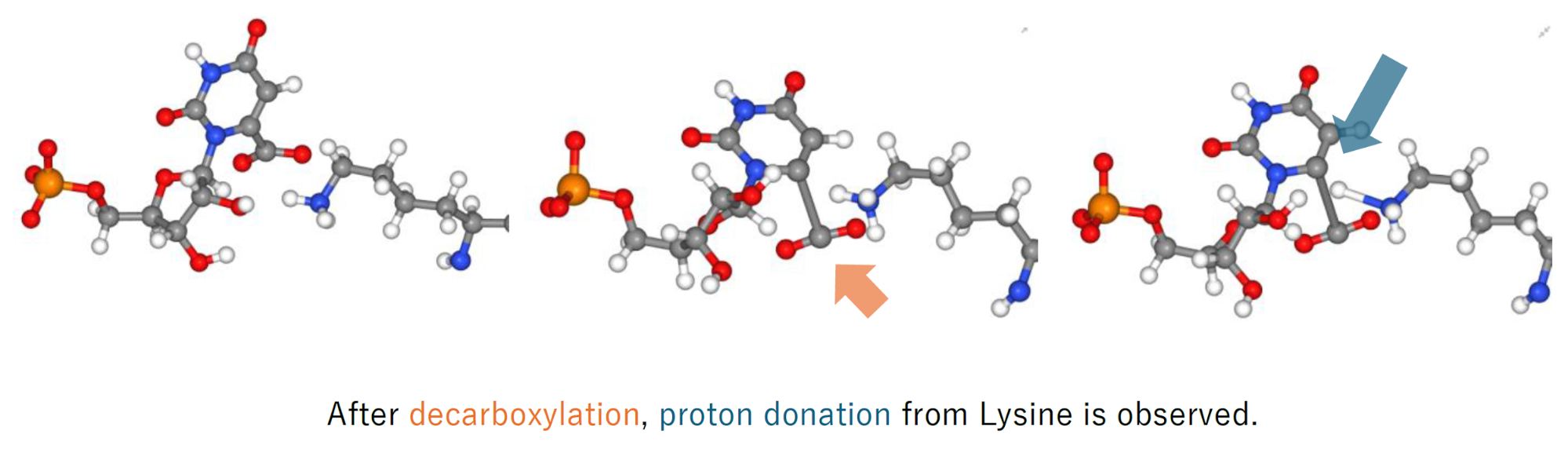
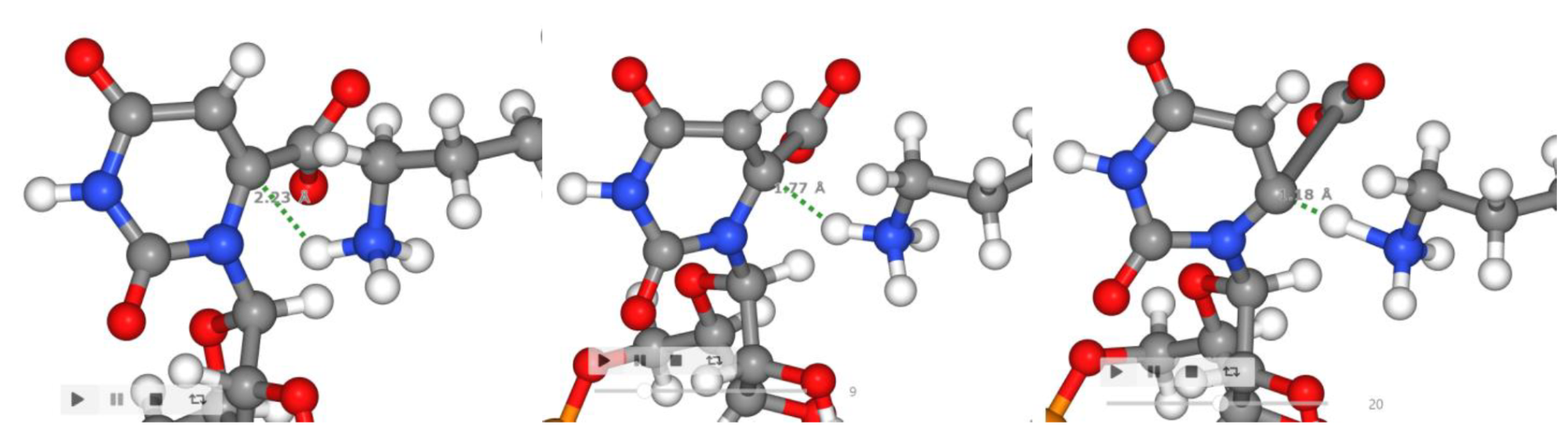
ここから酵素中での反応障壁を26.3 kcal/molと見積もりました。参考文献[1]にある実験値が17 kcal/mol、DFTによる計算結果が33 kcal/molであり、参考文献よりも実験値に近い結果となりました。また、水溶液中よりも酵素中の反応障壁が低く、酵素の触媒的な寄与を確認することができました。加えて下図のように、リシンからの水素原子の供与が確認されました。
LightPFPによる酵素中での反応障壁の計算
今回のインターンでは時間的な制約もあり、LightPFPによる反応経路は十分に収束したものではありませんが、現段階で得られたデータが以下のようになります。
カットオフを5 Åとして全系を学習データとして用いた場合、酵素中での反応経路は下図のようになりました。ここから得られた反応障壁の値は55.3 kcal/molとなり、PFPでの値とは大きく異なる結果となりました。
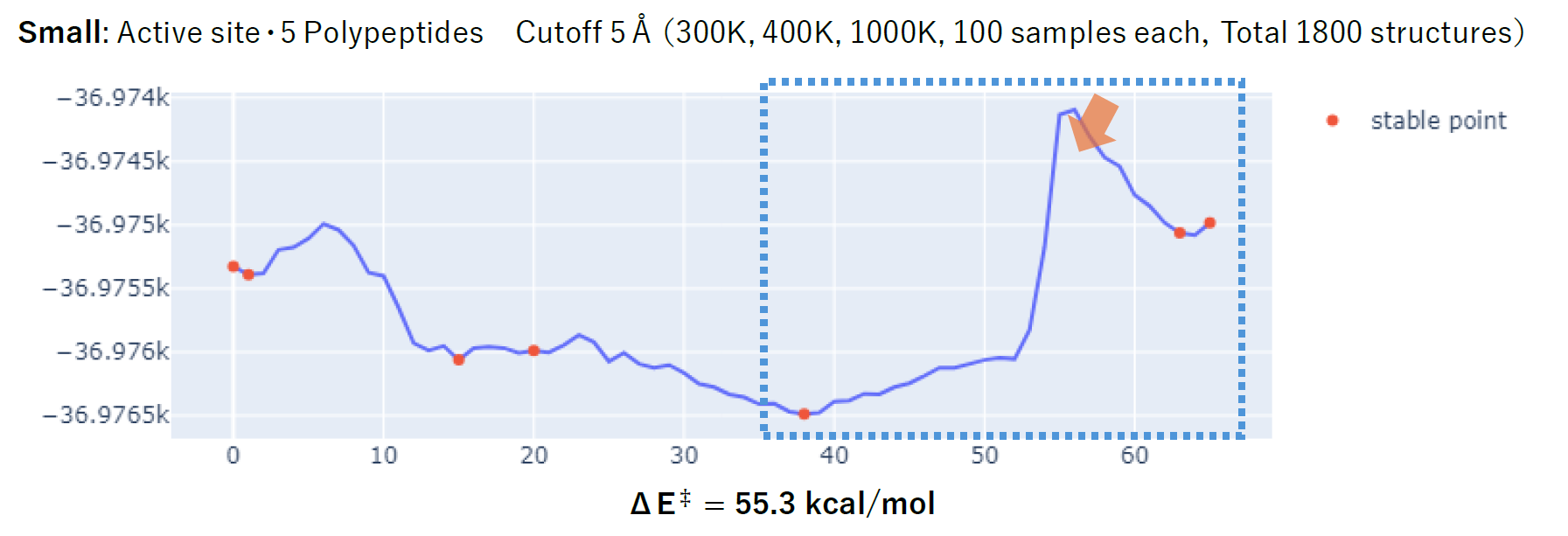
他のカットオフの値(10 Å, 15 Å)でも、全系を学習データとして用いた場合は、反応障壁の値に関して同様の結果となりました。加えてOMP分子の構造が壊れたり、リシンからの水素原子の供与が確認できず、定性的にもPFPの結果を再現できないことがわかりました。
一方で活性部位を選択的に学習データとして用いた場合、下図の反応経路から見積もった反応障壁の値は20.3 kcal/molとなり、PFPでの計算結果に近い値が得られました。
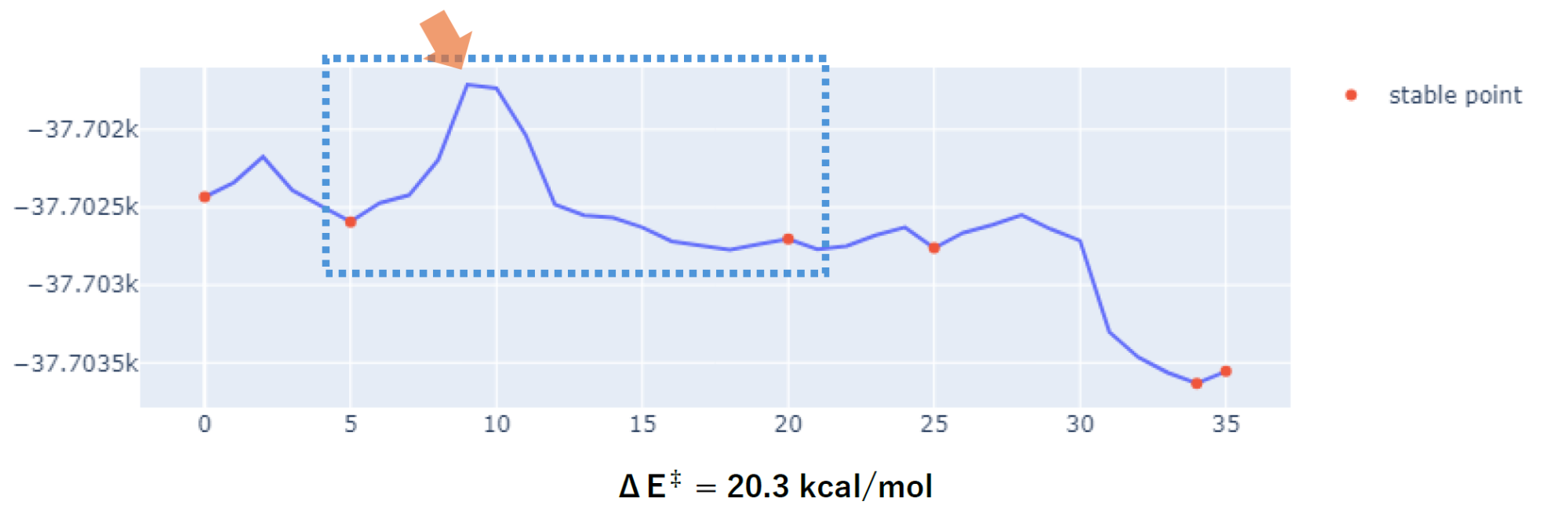
また下図のようにリシンからの水素原子の供与が確認でき、定性的にPFPでの振る舞いを再現することがわかりました。
まとめ・展望
LightPFPの学習には全系を用い、かつ系を切り取る際のカットオフ半径が大きい方が、PFPでの反応障壁の値をより再現できる、というのが当初の仮説でした。しかし今回の検証から、実際には学習データを反応部位のみに絞った方がPFPの結果をより再現できる、という結果が得られました。この要因としては、全系を学習に用いた場合は反応に関わらないペプチド鎖の原子がデータセットの内のかなりの割合を占めるため、反応に関わる箇所の学習がうまくできなかったことが考えられます。
また時間的な制約もあり、LightPFPの学習および反応経路の収束のために十分な時間が取れませんでした。現段階でも水素原子の供与の有無など定性的に議論できる部分はありますが、対象の系に対してより詳細な議論をするためには、LightPFPモデルをより頑健に訓練し、また経路の収束のために十分な時間をかける必要があります。
今回はLightPFPを用いた酵素反応の計算のための手法開発が主眼だったため、脱炭酸反応という比較的簡単な反応、かつPFPでも扱えるサイズの系を対象としました。今後の展望として、メチル基転移反応や多段階酵素反応といった複雑な反応や、PFPでは扱えないような大規模な系に対する応用が考えられます。
またLightPFPに関しては、今回の検証をMatlantis利用者向けのサンプルコードとして製品上で提供することで、酵素反応を扱いたい利用者をサポートすることができると思います。また、ポリペプチド鎖に特化したLightPFPモデルの事前学習済みモデルなどがあると、酵素に限らずタンパク質についての計算を行う際に役立つと思われます。
終わりに
本インターンではメンターの新谷さんを始めとする、Core-devチーム・chemチームの皆さんに大変お世話になりました。テーマの決定から研究方針の決定まで、十分に時間をとって議論していただくことができたうえ、不明な点にはすぐに答えていただけたので、2か月という短い時間でしたが満足のいく成果を得ることができました。
また、自身が日頃研究している酵素反応や機械学習ポテンシャルに近い分野でのインターンだったため、非常に多くの専門的な知識や経験を得ることができました。さらに機械学習ポテンシャルで酵素反応を扱うことができることをインターンで確認することができ、機械学習ポテンシャルの可能性を実感することができました。
最後に、インターン期間は短かったですが、それだけ集中して課題に取り組むことができたため、常に密度の高い充実したものとなりました。それに伴い自身のスキルの成長も実感しています。
これからも自身の研究も頑張っていきたいと思います!ありがとうございました。
参考文献
[1] J. Phys. Chem. B, Vol. 111, No. 43, 2007
[2] https://www.rcsb.org/structure/1DQX
Tag