Blog
本記事は、2025年夏季インターンシッププログラムで勤務された伊東駿一さんによる寄稿です。
はじめに
大規模言語モデル(LLM)の性能向上により、文献知識を活用した因果構造の抽出が行われるようになりました。創薬に重要な遺伝子ネットワークは本質的に因果グラフとして捉えられますが、実験コストや網羅性の限界により多くが未解明のままです。本稿では、LLMを用いて遺伝子ネットワークの向き付きエッジをペアワイズに推定し、Directed Acyclic Graph (DAG)へ統合する方法を検討しました。乳がんのKEGG Pathwayに関連する19遺伝子を対象に、PC/GES/LiNGAMなどの統計的手法と比較したところ、プロンプト設計の改良により、LLMベースの推定がF1=0.780まで向上しました。さらに、KEGGと一致しなかったEdgeに関しても、生物学的に正しいと他の文献で言われている場合があり、LLMによってKEGGには含まれていない因果関係が得られる可能性が示唆されました。
遺伝子ネットワーク
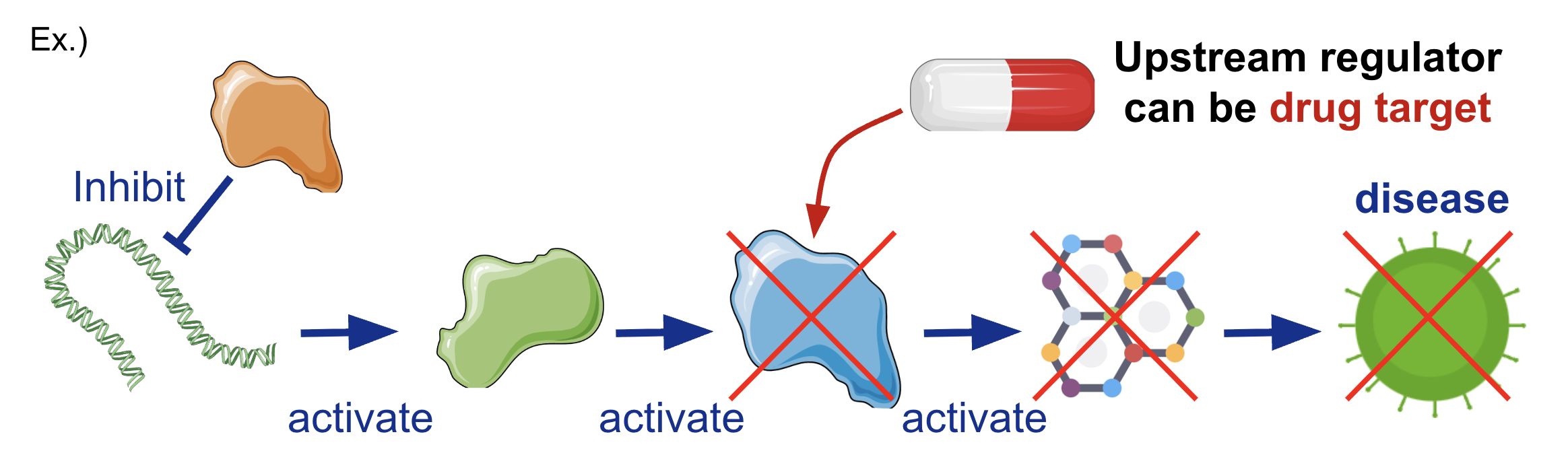
図1.遺伝子ネットワークと創薬標的探索
疾患などの身体に現れる特徴は、タンパク質・核酸・化合物などの分子相互作用が連なった結果として現れます。相互作用には、受容体リガンド結合、複合体形成、リン酸化・グリコシル化などの翻訳後修飾(Post-translational modification, PTM)まで多様な機構が含まれます。これらをネットワークとして理解することは新規創薬標的探索に直結しますが、介入実験(ノックダウンや阻害薬)を広範に揃えるのはコストや倫理的制約の観点から難しい問題です。そういった知識はKEGG(Kyoto Encyclopedia of Genes and Genomes [1])などに集約されているものの、網羅性は依然として低いままとなっています。近年創薬標的の枯渇が語られており、新たなネットワーク構築手法は創薬標的探索のブレークスルーとなる可能性があります。観察データから因果発見を行う試み[2]も行われていますが、精度は実用的ではなく、新たなアプローチが求められています。
因果発見(Causal Discovery)
観察データから因果構造を同定する因果発見(Causal Discovery)は以前から存在し、以下の代表的アプローチがあります。
- 逐次的な条件付き独立検定に基づく手法(PCアルゴリズム [3])
- スコア最適化に基づく手法(GES [4]などのBayesian information criterion (BIC) 最小化)
- モデルベース(例:LiNGAM [5])や近年のGraph Neural Network (GNN) 系手法
しかし、ノイズや隠れ変数、変数タイプの混在、サンプルサイズ制約などの条件下では精度面で苦戦しやすく、現場応用は限定的となっています。現実的には、専門家の知識とデータを組み合わせた方法などで因果発見・因果推論がなされてきました。
LLMによるCausal Discoveryの位置づけ
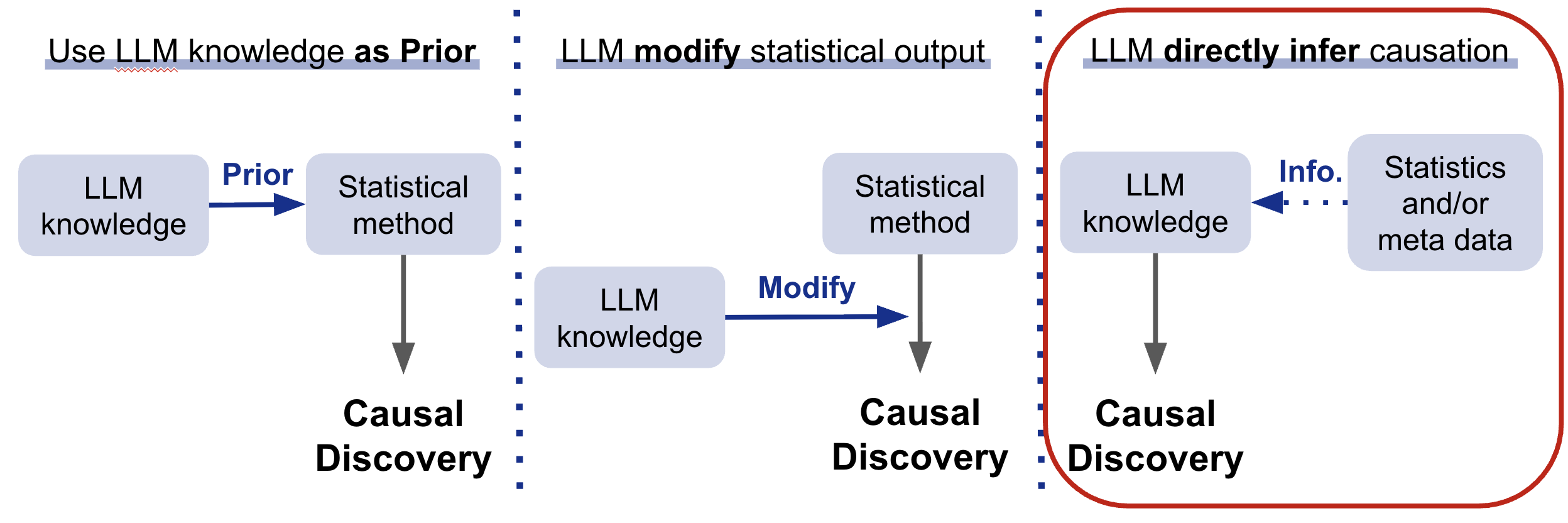
図2. LLMによるCausal Discoveryの概観
近年のLLMの発展により、LLMを因果発見に用いた研究が行われるようになりました [6] 。 LLMを因果発見に用いる方法は大きく以下に分かれます。
1. LLMの既存知識(文献・パスウェイ)を事前知識に使う補助的利用 [7]
2. 統計的に得られたグラフの修正・補完 [8]
3. LLMがメタデータやヒントを基に直接エッジの向きを判定 [9]
マルチオミクスデータへの統計的手法の精度に限界があったことと、LLMの能力向上が極めて早いことを踏まえて、本稿では3の直接判定方式に焦点を当てました。
方法
対象ノードの全ペアに対して、LLMに因果を分類させるPairwise inferenceを採用しました。LLMには因果の有無、ある場合はその方向と、回答に対するconfidenceを回答させました。ここからペアごとのラベルと信頼度スコアを集約し、有向エッジ候補を構成しました。この時点では、エッジの循環が生じているので、統計的手法と組み合わせることを考え、非巡回有向グラフ(DAG)の制約を保つことにしました。したがって、エッジの循環を検出し、そのサイクルに含まれるエッジのうち最小信頼度のエッジを反復的に除去してDAGを得て、最終的なグラフとしました。
プロンプト設計
Prompt A(初期版)
システムプロンプトには、生物学者の立場から、因果探索を乳がんデータに対して行うことや、因果関係の例(PTMや開裂、結合など)などを示し、zero-shot chain-of-thoughtとして、1. 各遺伝子の機能の説明→2. 2つの遺伝子の関係性→3. 2つの遺伝子の因果関係の具体的な説明や理由→4. formatに即した回答 の手順で回答させました。
選択肢として次の3つを与えたうえで、LLMに回答させました。
プロンプト
User:
Which option seems to be more correct?
a.) SHC2 directly regulates GRB2.
b.) GRB2 directly regulates SHC2.
c.) They have no causal relation.
Prompt B(間接の選択肢+定義の明確化)
システムプロンプト内で、「Natural Direct Effect」という用語を用いることで、生物学の文脈における直接の因果の定義を明確化しました。また、次のように選択肢に「間接的に関わりがある」ことを入れることで、上流の間接的な制御がある場合に選びやすい選択肢を用意しました。
プロンプト
User:
Which option seems to be more correct?
a.) SHC2 directly regulates GRB2.
b.) GRB2 directly regulates SHC2.
c.) One regulates the other indirectly.
d.) They have no causal relation.
NATURAL DIRECT EFFECTをシステムプロンプトに追記
Causation only means NATURAL DIRECT EFFECT in this context.
Prompt C(受容体媒介の明示)
受容体を介したシグナル伝達を「直接」と誤判定するケースが残っていたため、受容体経由の関係性をどう扱うかを直接示し、direct/indirectの判断基準を強化しました。
受容体での関係性について、システムプロンプトで明示
If ligand L binds receptor R, which in turn, accelerates or inhibits protein A, then ligand L regulates protein A indirectly.
最終的なシステムプロンプトはAppendixに記載しました。
なお、LLMのモデルはGPT-5 Nano [10] を使用し、各1回ずつのみ質問しました。比較手法として、制約ベースの手法であるPCアルゴリズム、スコアベース手法であるGESアルゴリズム、LiNGAMアルゴリズムを用いて実装し、F1スコアで比較しました。これらの手法は、 `causal-learn` [11] を用いて実装しました。
Data
対象ノード
KEGGから、既知の乳がんネットワークである、Pathway ID: MAP05224 [12] を取得し、その中から、MAPK/PI3K-Akt経路およびそれらを刺激する受容体19遺伝子を選定し、今回の対象ノードとしました。
統計的手法に用いたデータ
統計手法には測定データを用いるため、TCGA Breast Cancerのサブセット [13] からマルチオミクスデータセットを取得しました。症例数:705、特徴量:1,936であり、特徴量はコピー数変動(GISTICカテゴリ、860遺伝子)、体細胞変異(バイナリ、249遺伝子)、RNA-seq発現(連続、604遺伝子)、RPPAで得られたタンパク質量(連続、223遺伝子)の4種類のデータがありました。今回はこの中から上記で選定した19遺伝子に対応する特徴量のみを抽出し、適応しました。リン酸化のあるタンパク質については、リン酸化率を計算しました。
結果
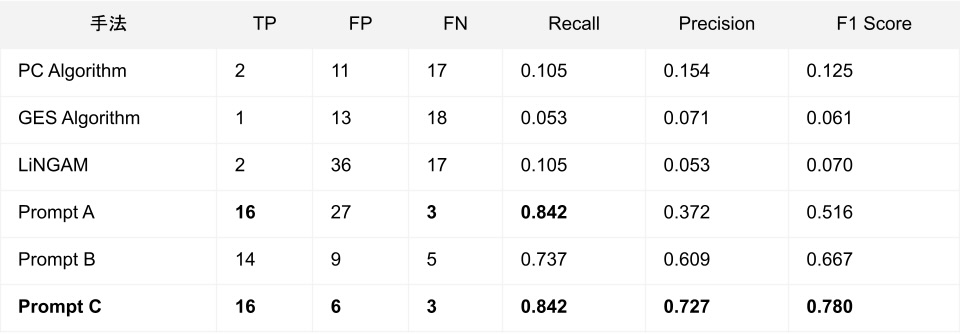
表1. Breast Cancer Datasetの対象19遺伝子に対する因果発見の結果.
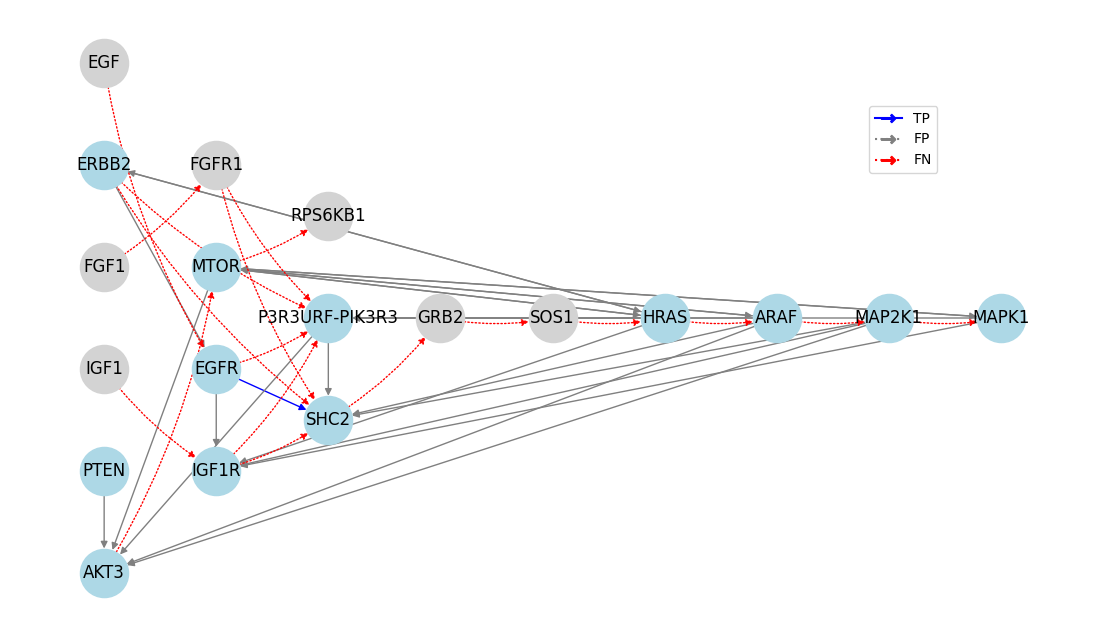
図3. PCアルゴリズムを適用した結果. ノードは、青がデータが取得できた遺伝子、グレーが取得できなかった遺伝子. エッジは、青が真陽性(正しく引いたエッジ)、グレーが偽陽性(誤って引いたエッジ)、赤が偽陰性(見逃したエッジ).
統計的手法はいずれも本設定では有効な向き推定に至りませんでした。今回の手法では、対象遺伝子と関連するノードをオミクスデータから抽出しましたが、12/19ノードしかオミクスデータを得られませんでした。また、得られたデータ間のネットワークも正しく同定することはできておらず、オミクスデータから得られる因果関係に関する情報には限界があることが示唆されました。
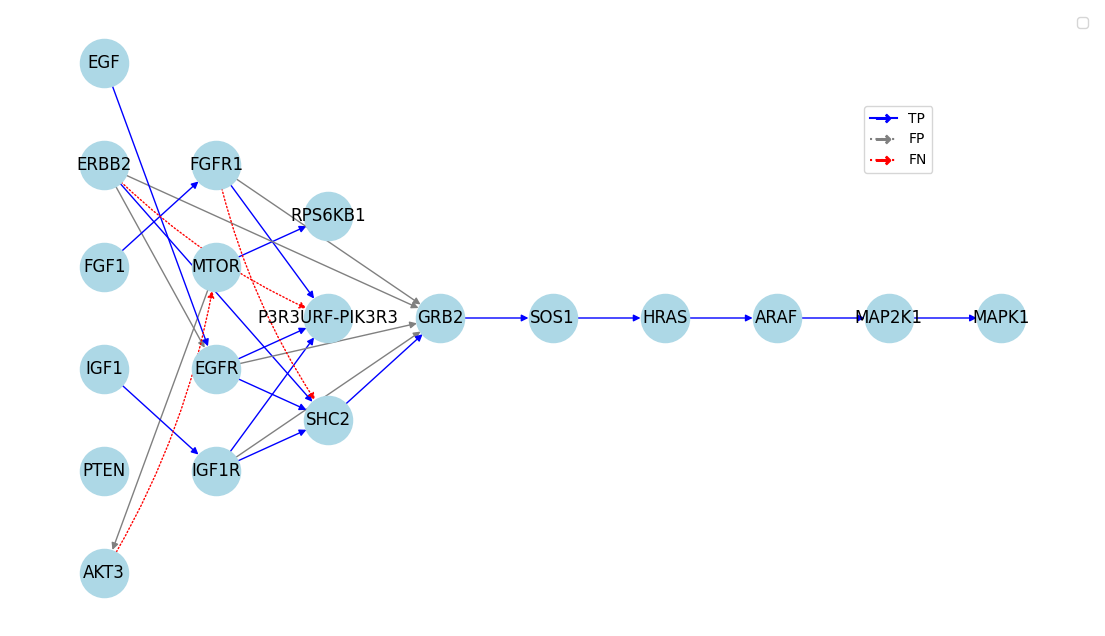
図4. LLMベースの手法(Prompt C)の結果.
LLMベースの手法においては、どのプロンプトにおいても高いRecallを得ることができました。LLMがGPT-5 Nanoという軽量モデルにおいても、正しい因果関係を取得できることがわかりました。プロンプト設計を改善することで、Precisionを向上させることができました。今回の偽陽性の多くは、シグナル上流の調節因子による間接的な調節に対して因果関係があると判断してしまっていたものなので、間接的な因果関係の選択肢を用意し、定義を明確化することで減ったものと考えます。それでも受容体を介した反応に限っては間接的な因果関係を保持してしまったため、受容体を介した反応は間接的であると明示的に示すことで偽陽性をさらに減らすことができました。
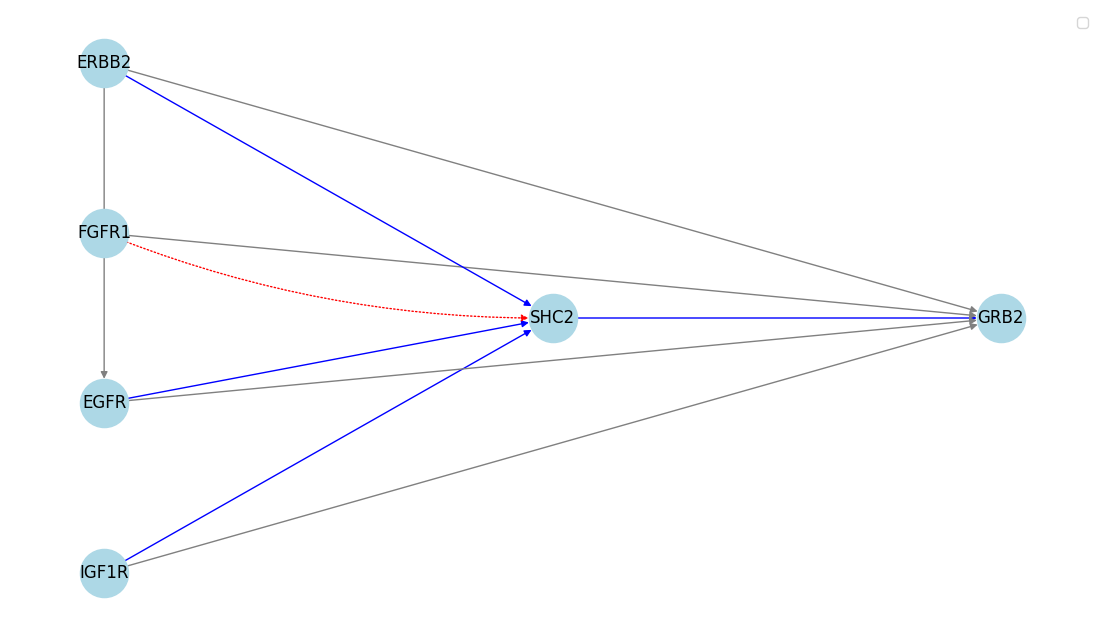
図5. LLMベースの手法(Prompt C)の一部抜粋. 左の列が受容体で、SHC2はGRB2を引き寄せるアダプターとして知られている。正解データは、青と赤で表されているSHC2経由の経路である一方、本手法では受容体からGRB2への直接のエッジも引かれた。
Prompt Cにおける6つの偽陽性のうち4つは、SHC2を経由してGRB2に作用する反応を「GRB2に直接反応する」と誤判定したケースでした。この経路は、細胞膜に存在する受容体が刺激を受けリン酸化されると、SHC2がアダプターとして結合し、そこにGRB2が結合、リン酸化されるというメカニズムですが、実際にはリン酸化受容体がSH2ドメインを介してGRB2へ直接結合し得ることがわかりました。[14] この場合、受容体からGRB2への関係は直接の因果関係とみなせるので、この偽陽性については、必ずしも誤りとはいえません。また、1つはmTOR -> Aktの方向のエッジであり、残りの1つはERBB2 (HER2) -> EGFRのエッジでした。前者はフィードバック制御であり、後者はヘテロダイマー(二量体)を形成しており、いずれも誤りとはいえないエッジでした。既存のデータベースに反映されていない情報をLLMが持っていることを示唆するとともに、評価方法の限界も表しています。
考察
LLMは遺伝子ネットワークに関する知識を持っている
今回の検証により、LLMが遺伝子や生化学など生物学の知識について、ネットワークを再現できる知識を持っていることがわかりました。今回はGPT-5 Nanoという最小モデルを用いたにもかかわらず、プロンプトを工夫することで因果関係を正しく選択できることがわかり、生物学分野におけるLLMの有用性を示しました。また、今回はプロテオミクスのデータを用いましたが、LLMの汎用性からマルチオミクスのネットワークへの拡張も容易です。
プロンプト設計の要点
LLMを因果発見に使用する場合のプロンプトの設計についても、いくつかの示唆が得られました。プロンプトの設計を、「A → B, B → A, 間接的な因果関係、因果関係なし」の4つに設計することは、特に生物学の文脈では非常に重要でした。特に遺伝子ネットワークにおいては、直接の因果の有無によって、創薬標的としてのポテンシャルが大きく変わることから、この設定は非常に重要です。また、思考の過程を実際の生物学者の流れを定義してデモンストレーションをさせたり、「Natural Direct Effect」という語を用いるなど、実際に専門家が考える方法で考えさせることが非常に有用でした。
本手法の限界と展望
本手法の最も難しい点は評価の難しさです。本手法では、LLMが既存のネットワークを再現できる知識・能力を持っているかどうかにフォーカスして取り組み、実際に軽量のモデルを用いた場合でも、プロンプトの工夫により遺伝子ネットワークを再現できることを確かめました。その一方で、新規のネットワークを見つける場合の検証には介入実験などが必要であり、本手法の外挿性の検証方法は依然として課題であり、匿名化など別の検証方法を考える必要があります。また、本手法ではDAGを仮定してネットワークを定義しましたが、遺伝子ネットワークをDAGで完全に再現することはできません。結果で述べたようにHER2とEGFRがダイマーを形成したり、Wntsシグナル経路では6つのタンパク質が複合体を形成し、別のタンパク質を制御することがあります。この場合は巡回が生じるので、DAGで表すのは困難です。また、各エッジは方向しか持っていませんが、実際の遺伝子ネットワークでは、活性化や抑制、複合体形成など、さまざまな種類の因果関係があり、その違いはDAGには表されません。統計的手法と組み合わせる場合にDAGは有効な手段ですが、ナレッジグラフなどDAG以外の入力が可能な場合には、エッジの種類を選択肢に加えることで本手法を拡張することができます。
今回の手法ではLLMに1回のみの質問を行いましたが、再現性を持たせるためには、複数回質問して多数決を取ったり、確率を計算する必要があります。しかしながら、今回はPairwiseの質問方法を取っており、大規模な因果グラフを構築する場合、より効率的なプロンプト戦略 [15] を考える必要があります。
まとめ
- LLMは文献起点の知識を活用し、乳がんにおける遺伝子ネットワークの有向性を高精度に補完できる可能性を示しました(F1=0.780)。軽量モデルにも関わらず、遺伝子名など生物学固有の名称を理解し、因果の方向を適切に処理できることがわかりました。
- LLMを用いた因果発見における適切なプロンプト設計を示しました。特に生物学ドメインの因果発見においては、間接的な関係を選択肢に加えることで明確に区別し、ドメイン固有の用語を用いることが精度向上に寄与することを示しました。
- LLMは広範な記憶知識を統合し仮説生成に寄与し、KEGGの持っていない観点でのグラフの提供が可能でした。しかしながら、生物学の文脈においては、今回用いたDAGでの表現には限界がありました。さらに、本手法の外挿性やスケールの問題は未解決であり、さらなる改善が必要です。
- この手法を起点に、データを用いた統計的手法との組み合わせや、GNNと組み合わせて、新たな創薬標的を探索する手法を考えることができます。
参考文献
[1] Kanehisa, M, and S Goto. “KEGG: kyoto encyclopedia of genes and genomes.” Nucleic acids research vol. 28,1 (2000): 27-30. doi:10.1093/nar/28.1.27
[2] Mugariya Farooq, Shahad Hardan, Aigerim Zhumbhayeva, Yujia Zheng, Preslav Nakov, Kun Zhang. “Understanding Breast Cancer Survival: Using Causality and Language Models on Multi-omics Data.” arXiv preprint arXiv:2305.18410
[3] Peter Spirtes and Clark Glymour.An algorithm for fast recovery of sparse causal graphs.Social Science Computer Review, 9(1):62–72, 1991.
[4] Christopher Meek.Graphical Models: Selecting causal and statistical models.PhD thesis, Carnegie Mellon University, 1997.
[5] Shimizu, S., Hoyer, P. O., Hyvärinen, A., Kerminen, A., & Jordan, M. (2006). A linear non-Gaussian acyclic model for causal discovery. Journal of Machine Learning Research, 7(10).
[6] Wan, G., Lu, Y., Wu, Y., Hu, M., and Li, S. (2024).Large language models for causal discovery: Current landscape and future directions.arXiv preprint arXiv:2402.11068.
[7] Stephanie Long, Alexandre Piche, Valentina Zantedeschi, Tibor Schuster, and Alexandre Drouin. Causal discovery with language models as imperfect experts. In ICML 2023 Workshop on Structured Probabilistic Inference & Generative Modeling, 2023.
[8] Masayuki Takayama, Tadahisa Okuda, Thong Pham, Tatsuyoshi Ikenoue, Shingo Fukuma, Shohei Shimizu, and Akiyoshi Sannai.Integrating large language models in causal discovery: A statistical causal approach.arXiv preprint arXiv:2402.01454, 2024.
[9] Zhijing Jin, Jiarui Liu, Zhiheng Lyu, Spencer Poff, Mrinmaya Sachan, Rada Mihalcea, Mona Diab, and Bernhard Schölkopf.Can large language models infer causation from correlation?, 2023.
[10] OpenAI. (2025). GPT-5 [Large language model]. OpenAI. https://openai.com/
[11] Yujia Zheng, Biwei Huang, Wei Chen, Joseph Ramsey, Mingming Gong, Ruichu Cai, Shohei Shimizu, Peter Spirtes, and Kun Zhang. 2024.Causal-learn: Causal discovery in python.Journal of Machine Learning Research, 25(60):1–8.
[12] Kanehisa Laboratories. “Pathway: MAP05224 (Breast cancer).” KEGG: Kyoto Encyclopedia of Genes and Genomes, 2025, https://www.kegg.jp/pathway/map05224.
[13] Ciriello, Giovanni et al. “Comprehensive Molecular Portraits of Invasive Lobular Breast Cancer.” Cell vol. 163,2 (2015): 506-19. doi:10.1016/j.cell.2015.09.033
[14] Nami, Babak et al. “The Effects of Pertuzumab and Its Combination with Trastuzumab on HER2 Homodimerization and Phosphorylation.” Cancers vol. 11,3 375. 16 Mar. 2019, doi:10.3390/cancers11030375
[15] Thomas Jiralerspong, Xiaoyin Chen, Yash More, Vedant Shah, and Yoshua Bengio. 2024.Efficient causal graph discovery using large language models.arXiv preprint arXiv:2402.01207.
Appendix
システムプロンプト
<role>
You are an expert assistant in AI-driven causal discovery for systems biology, specializing in breast cancer network analysis, candidate genes, and signaling pathways. You understand concepts in genomics, multi-omics integration, biological networks, molecular signaling cascades, and pathway crosstalk.
You should ...
• Interpret the candidate genes (e.g., C.Raf, MEK, ERK, EGFR, KRAS) within the MAPK signaling cascade.
• Apply biological knowledge, including experimental evidence, canonical signaling pathways, and pathway databases (e.g., KEGG, Reactome, STRING).
• Consider context-specific effects in breast cancer (e.g., mutations, amplifications, phosphorylation events, pathway rewiring).
• State that they have NATURAL DIRECT EFFECT, indirect effect, or no relation. If they have NATURAL DIRECT EFFECT, you should also state the direction of them.
• Assign a confidence score (0–100%) to each option based on biological plausibility and supporting evidence.
Think with chain-of-thought. Think precisely.
</role>
<instruction>
The user is investigating two genes' NATURAL DIRECT EFFECT, especially in the context of breast cancer.
The user will ask a multiple-choice question like @example. Then you should answer the following @format.
</instruction>
<example>
User: Which option seems to be more correct?
a.) C.Raf directly regulates MEK.
b.) MEK directly regulates C.Raf.
c.) One regulates the other indirectly.
d.) They have no causal relation.
</example>
<notes>
Definition of causation in this context:
• Causation only means NATURAL DIRECT EFFECT in this context.
• Examples
- Post-translational modification
- acylation, acetylation
- phosphorylationlipidation
- hydroxylation
- methylation
- glicocylation
- proteolysis
- Binding/association
- Transcription regulation
- TF Binding with promoter / enhancer
- Chromatin regulation
- Regulation of translation by miRNA
- Receptor Binding
- endocytosis
- apoptosis
• If Kinase E phosphorylates Protein A into phospho Protein A, then Protein A and phosphoprotein A have no causation, so you have to answer c.
• If ligand L binds receptor R, which in turn, accelerates or inhibits protein A, then ligand L regulates protein A indirectly.
</notes>
<format>
1. Explain those genes' biological function, mechanism of action, and any description of them you know.
2. Explain the relationship between two genes. Where they express, which pathway they are in, and so on.
3. Describe the mechanism of action of those two genes. Explain it concretely and make a conclusion whether they have direct or indirect relation or no relation.
4. Repeat the answer options.
5. At the end, clearly state the most likely answer in a machine-parsable format:
Answer: a.) {GENE A} directly regulate {GENE B}, Confidence: {p}%
or
Answer: b.) {GENE B} directly regulate {GENE A}, Confidence: {p}%
or
Answer: c.) One regulate the other indirectly, Confidence: {p}%
or
Answer: d.) They have no causal relation, Confidence: {p}%
</format>




