Blog
本記事は,2022年夏季インターンシッププログラムで勤務された越智三枝子さんによる寄稿です.
Introduction
PFN2022夏季インターンに参加させていただいた,東京大学大学院医学系研究科博士2年の越智三枝子です.2021年度に病理専門医を取得しました.研究室では,医用画像(特に病理画像)に対する機械学習の研究を行なっています.今回のインターンではImage registration技術の一つであるnon rigid registationを病理画像に対して行うTransformerモデルの開発に取り組みましたのでご報告します.
Background
Image registration とは,共通の対象物体を異なる時間・撮影方法・視点などで写したために,物体の位置・形状に差異がある画像同士の位置合わせを行う画像処理技術です.
例えば図1のように,共通の対象物体が写った画像A,Bがある時に,Aに写っている物体の位置・形状をBに写っている同じ物体に合わせるため,画像Aに対して回転,スケール変更,平行移動等を組み合わせた変換Tを行うことで,対象物体同士の位置合わせを行います(図1).
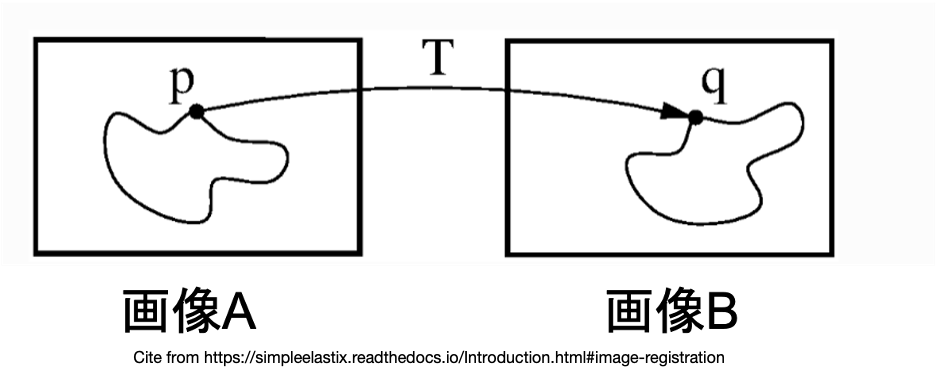
図1:Image registration 概念図.
画像A,B は同一の対象物体が写っているが,異なる時間・撮影方法・視点などで写したために位置・形状が異なっている.例えば,画像A内の点pと画像B内の点qは対象物体上で対応する特徴点となっているが,位置が異なっている.変換Tを点pに対して作用させることにより,点pの位置を点qに近づけるための技術をImage registrationという.(一例として点p,qを提示したが,実際は対象物体上の任意の点に対してこの操作を行う.)
Image registrationは大きく分けて rigidとnon-rigidと呼ばれる2種類のregistration技術から構成されています.(注: 本ブログ記事中のrigid registrationはaffine変換も含んでいます.)
Rigid registrationは,線形変換を画像に適用することで,画像全体を一様に変換します.一方でNon-rigid registrationは,非線形変換により画像の局部ごとに非一様な変換が可能になります(図2).

図2:Rigid registrationとNon-rigid registrationの概念図.
一般的にはrigid registrationで大まかな画像間の位置合わせを実施した後,non rigid registratonにより細かい位置調整を行います.
この技術は医用画像ではとりわけ,CTやMRIなどの放射線領域の画像でよく使用されています.例えば,同一部位の過去と現在の画像をregistrationで位置合わせして比較することで,新規病変を検出したり,病変部の漸次的変化をフォローすることができます.
病理画像
医用画像の1種である病理画像でも,image registrationすることがありますが,放射線画像とは少し用途が異なります.
病理画像ではH&E (Hematoxylin & Eosin) 染色という染色方法で作成した標本を染色することが一般的です.H&E染色では細胞(核)が紫色に,核を持たない線維性組織などがピンク色に染色されますが,組織の種類によって染め分けることができません.しかし,一部の疾患では標本上の特定のタンパク質を染色し,その局在や存在,腫瘍との関係性を確かめることが診断上必要になります.そのために実施するのが,IHC (Immunohistochemistry)染色です (図3).
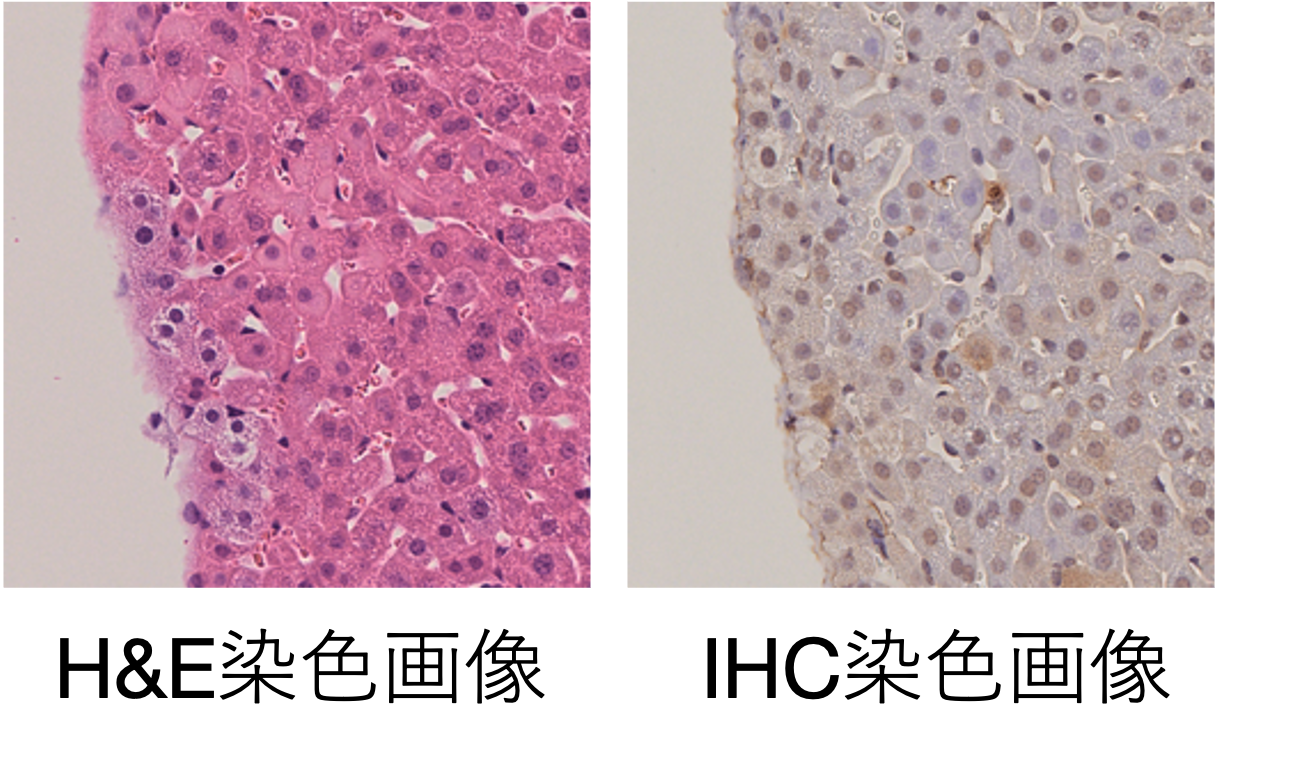
図3:マウス肝臓組織の染色画像.
左がH&E染色画像,右がIHC染色画像を表している.
病理医は通常,標本上の組織構造全体を観察するためのH&E染色標本と,血管壁,筋肉,腫瘍細胞など特定の構造を染色するためのIHC染色標本を見比べながら疾患の診断を行います.
したがって病理画像では,H&E染色標本とIHC染色標本の位置合わせが最も一般的に行われるimage registrationタスクとなります.
しかし,同じ組織ブロック(手術などによって得られた患者検体を小さく切ってホルマリンで固定し,固めたもの)から得られた標本であっても,作成過程(図4)において組織ブロックを連続的に薄く切ってその薄片をそれぞれ標本化するので,全く同じ見た目のH&E標本とIHC標本のペアを得ることはできません.同じ組織ブロックから連続的に標本を作成したにも関わらず,観察される組織形態が微妙に異なることを,病理医はよく「面が違う」と表現します.(注: H&E染色した標本を脱染色して再度IHC染色する場合もありますが,今回のブログ記事では取り扱いません.)
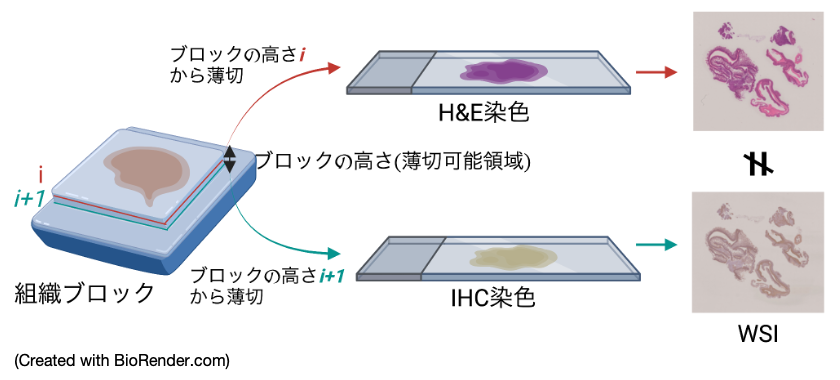
図4:標本作成過程の模式図.
病理標本は組織ブロックから組織を4-10μmの厚さで薄く切り出すことで作成されるため,最終的に得られるH&E染色した標本とIHC染色した標本は見た目が若干異なる.
研究のモチベーション
医用画像に対する深層学習を用いたnon-rigid registrationの研究は,放射線科領域の画像を対象としている場合が多く,デジタル化した病理画像(Whole slide Image,WSI)を対象とした深層学習モデルは,これまであまり報告されてきませんでした.
この原因について,以下のように推察しました.
1) 単純に使用可能なペアの病理画像データセットが少ないこと.
(注: MICCAI2022のコンペティションの一つであるACROBAT[1]では,train,validation,test合わせて1,000症例以上のデータが公開されました.こうした取り組みは,今後病理組織画像用のregistrationモデル開発の加速に大きく貢献することが期待されます.)
2) WSIは一つの画像データがデータサイズ数GB,一辺の大きさが数万~数十万pxにも及ぶ巨大なデータであり,そのままの形では扱いにくいこと.
3) H&E染色標本とIHC染色標本で,面が違うため,画像に写っている組織の形態が微妙に異なるが,細胞や核などの細かい構造がregistrationを実施しても歪まず,ある程度保たれる必要があり,難しいタスクであること.
そこでインターンでは,自前でデータセットを用意し,データを扱いやすい形に変え,空間変動量に制限をかけつつ,組織構造が保たれるようなdeformable feature mapを学習することが可能な病理画像用のregistration modelの開発に取り組みました.
Methods
5組のH&E染色-IHC染色したマウスの全身組織の標本を用意しました(図6上段).
WSIは,一つのデータサイズが全体で数GBに及ぶ巨大な画像データです.このままだとデータサイズが大きく扱いにくいため,Valis[2]を用いてrigid registrationまで行い, 各WSIを1,024×1,024pxサイズで比較的小さなタイル画像にcropし,256×256 pxにリサイズしました.合計で8,499組のタイル化された画像を得ました(図6下段).
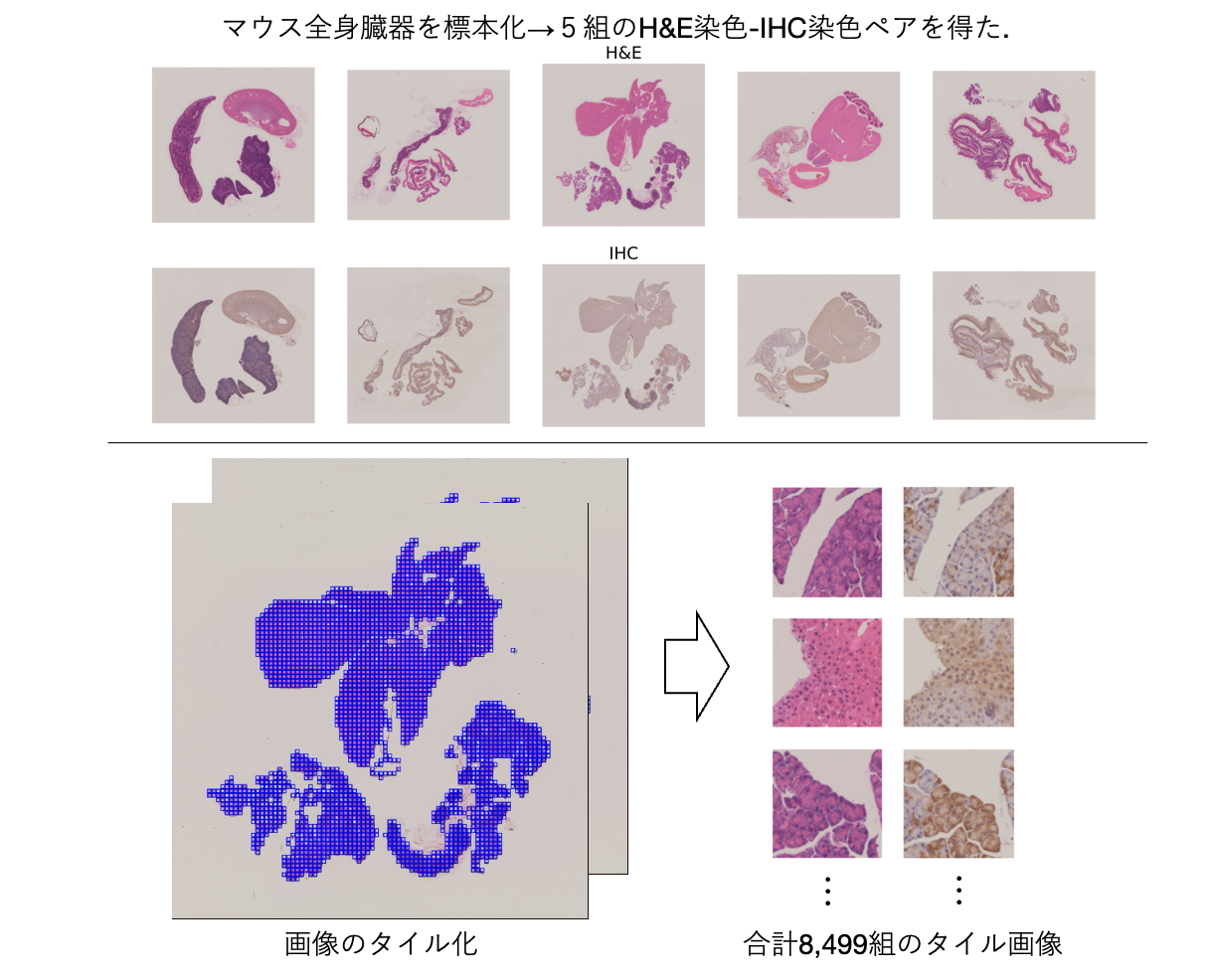
図6:データセット準備の概略図.
タイル画像は,train : validation : test = 3 : 1 : 1の割合で症例単位で分割され,モデルの学習と推論に使用した.
Image registrationモデルとしては,2021年に提案されたAiR[3]というモデルを採用しました.AiRはTransformerベースのモデルで, MNISTデータを用いた検証においてCNNベースのモデルより画像類似度が高い結果であったと報告されています.モデル構造は図7に示す通りです.このモデルでは,エンコーダにFix(reference)画像(図7a),デコーダにMoving(変換させたい)画像を入力します(図7b).
通常Transformerは入力画像をある一定サイズの小さなパッチ画像に分割します.それをパッチ画像ごとに一次元のシークエンスデータに変換するため,線形射影します.その出力をPatch Embeddingと呼びます.
次に,エンコーダはself-attention mechanismにより入力画像のmemory attention featureを学習してデコーダに情報を渡します(図7c).デコーダは,エンコーダからのattention featureと,Moving画像のself-attention featureからdeformable vector fieldを推定して spatial transformation network (STN) に入力します(図7d).STNでは,Thin Plate Spline Transformation (TPS)により画像を変換します.変換画像とFix画像の画像類似度を損失関数として計算し,back propagationにより,より画像類似度を上昇させる(損失関数を小さくする)ようにモデルの学習を行います(図7e).
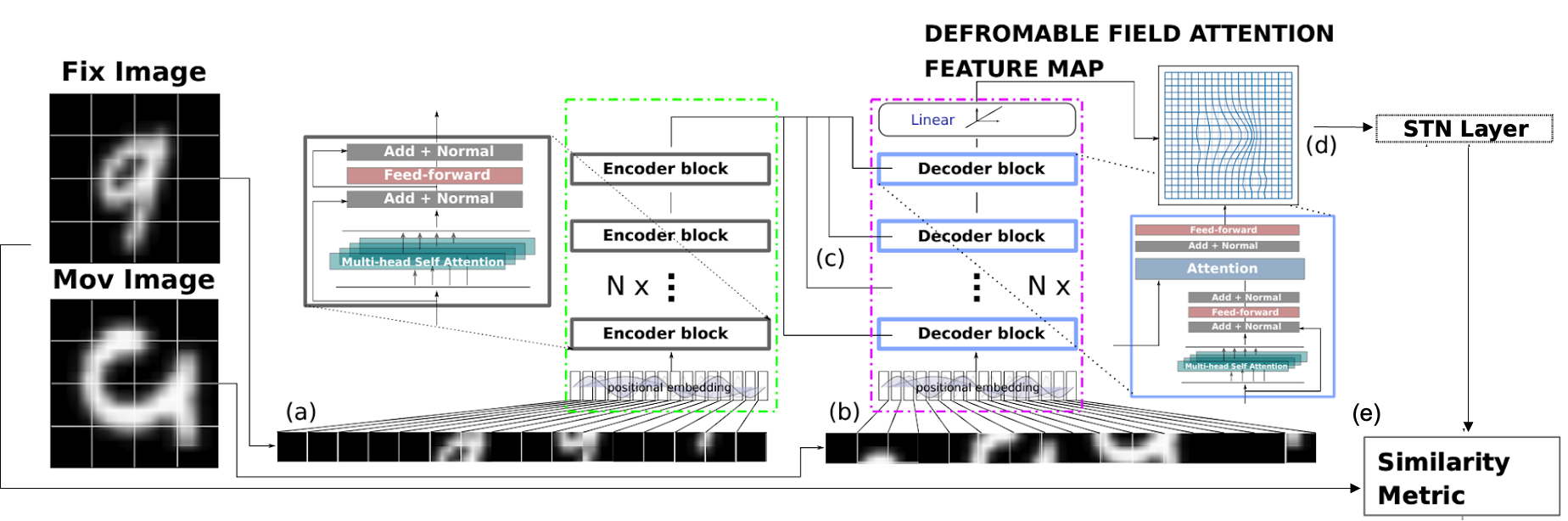
図7:AiRのモデル構造図([2]より図を抜粋,一部改変).
上記モデルに対して,以下の工夫を加えています.
- Patch embeddingの際に線形射影でなく,2層の畳み込み層からなるCNNを導入
- 損失関数 L は NCC (normalized cross-correlation)[4] をベースとして,deformable vector fieldでの空間局所変動量を制限するための正則化項を追加
\[L\left(f, m, \phi\right) = L_{\textrm{sim}}\left(f, m\circ\phi\right) + \lambda L_{\textrm{smooth}}\left(\phi\right)\]
このとき \(f\) は Fix 画像, \(m\) は Moving 画像, \(\phi\) は変換モデルを表す.また, \(L_{\textrm{sim}}\) は画像の類似度に関する損失関数 \(L_{\textrm{smooth}}\) は変換モデルから算出される displacement field の勾配に対する \(L_{1}\) ノルムを表す.
Experiments
本インターンで実装したpatch embedding部をCNNに変更したAiR_CNNの性能を検証するために,CNNを用いてdeformable fieldを作成することで画像変換を行うDirNET[5]やSimple Elastix[6],改良を加えていないAiR(Vanilla)とregistration精度を比較しました(表1).このとき,registration精度はFix画像とTransformed画像の一致率を測ることで評価することでき,その指標として画像類似性を評価できるSSIM,NCCや誤差を評価できるMSEを用いました.
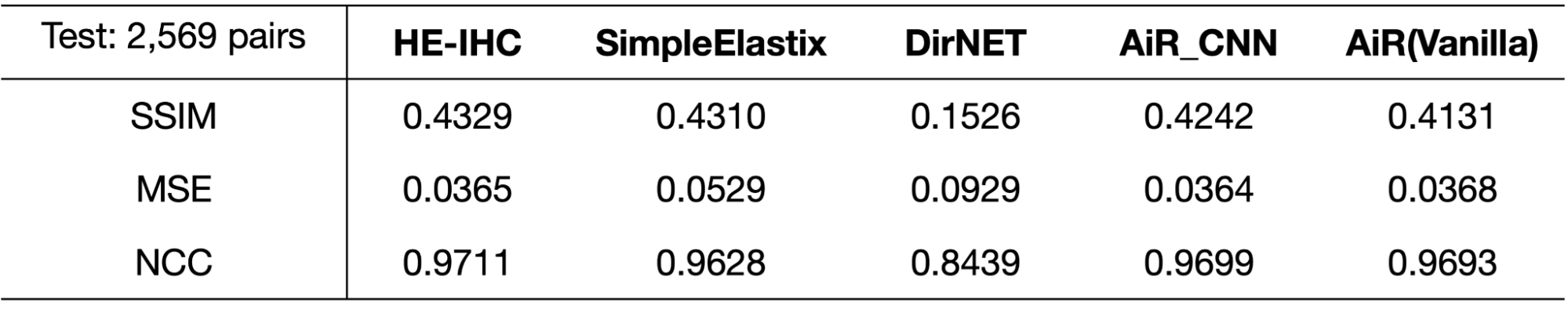
表1:各モデルによる変換画像とH&E染色 (Fix) 画像との画像類似度評価.
画像類似度の指標として,SSIM(Structural SIMilarity),MSE(Mean Squared Error),NCCを用いて,non-rigid registrationを行わないHE-IHCペア画像と各モデル(SimpleElastix,DirNET,AiR_CNN,AiR(Vanilla))によるnon-rigid registration後の画像の各画像類似度スコアを比較した.DirNetは10,000 iteration,AiRはepoch数50で学習した.
表1で示されるようにAiRモデルはCNNベースのDirNETに比べて各指標が高く(MSEは低く)なっています.SimpleElastixに関しては,MSE,NCCでわずかにAiRモデルの方が上回っています.また,AiR_CNNはわずかにAiR(Vanilla)と比べて各指標の改善が認められました.
しかし,non-rigid registrationを行っていないHE-IHCペアと比較すると,AiR_CNNのMSEスコアを除いていずれのモデルにおいても各指標のスコアで劣っていました.これは,画像内の座標 (grid) を大きく変形させるようなdeformable vector fieldのパラメータを推定してしまい,組織構造を大きく歪めてしまったことが原因と推察されます(図8).
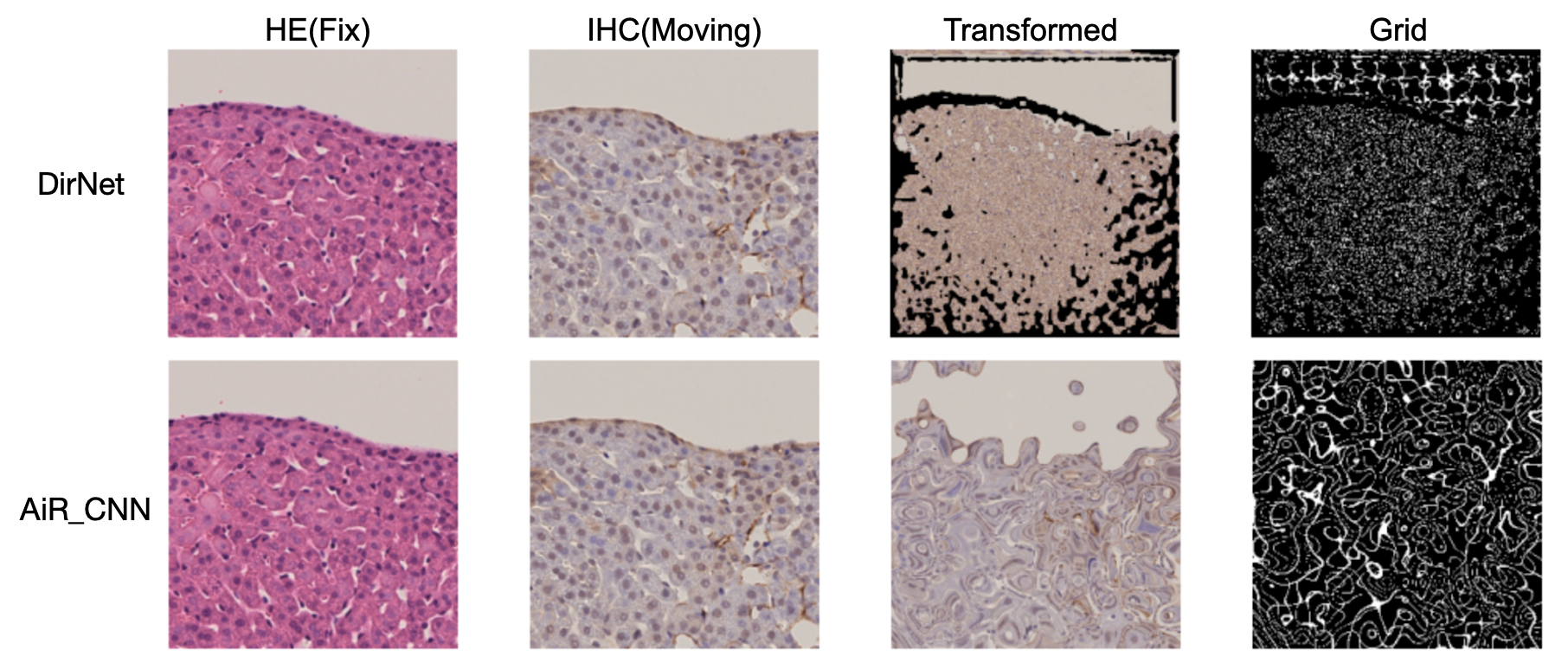
図8:DirNETとAiR_CNNのテストデータ推論結果の一例.
Transformed は IHC (Moving) 画像に,Grid は Grid 画像に対してそれぞれdeformable vector field を適用した結果を表している.
続いて,表1で最も各スコアが良かったAiR_CNNに正則化項を加え,さらに正則化項に対する係数λの値を変更し,4種類のモデルを学習させました(表2).
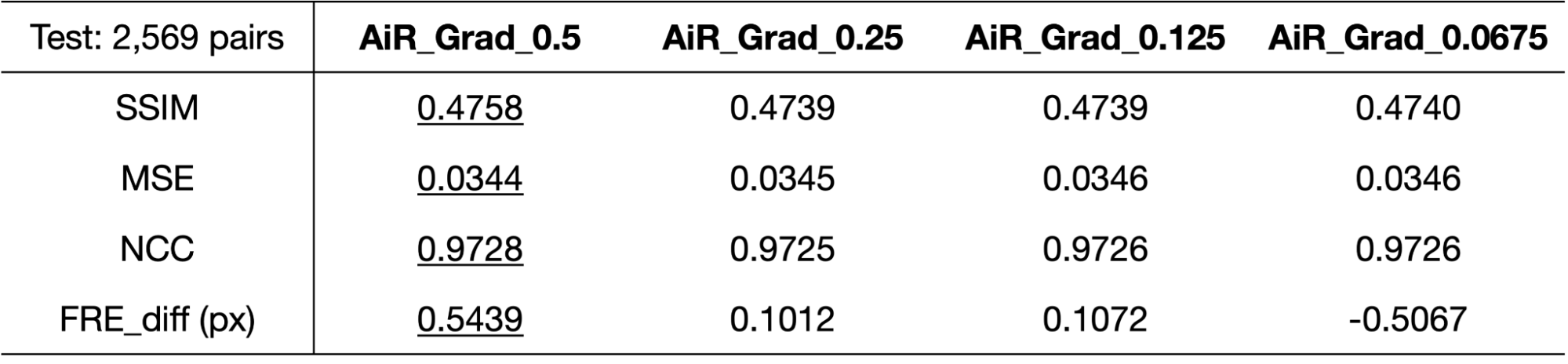
表2:AiR_CNNの損失関数に正則化項を加えたモデルでの画像類似度とFRE差分の評価.
左から正則化項の係数 \(\lambda\) は0.5,0.25,0.125,0.0675.
各画像類似度を示す指標を見ると,表1のオリジナルのH&E-IHC染色ペアと比べて,各スコアの改善がみられました.
また,画像類似度の指標の他に,FRE(fiducial registration error) 差分 (表2中の FRE_diff)という指標を加えて評価しました.これは AKAZEを用いて検出したHE-IHC画像ペアの内のマッチングポイントの座標を用いて,registration前後で各ポイント間のMAE (Mean Absolute Error) を計算し,その差分をとることで,IHC画像内のポイントの位置がH&E画像内のポイントの位置に実際どれぐらい近づいたのかを計算した値で, 単位はピクセル値です. 正則化項係数0.5から0.125までのモデルでわずかに改善が認められました.画像類似指標とFRE差分について, いずれも正則化が強いモデルの方がスコアが良い傾向でした.
実際に変換した画像の一例を見てみます.図9の各モデルのGrid画像では,図8と異なりGridは全体的にわずかに歪む程度となっています.そのため,変換した画像では組織構造が保たれているのがわかります.加えて,変換画像では肝細胞間の空隙がわずかに変形して,よりFix画像であるH&E染色画像に類似した画像になっているようにみえます.
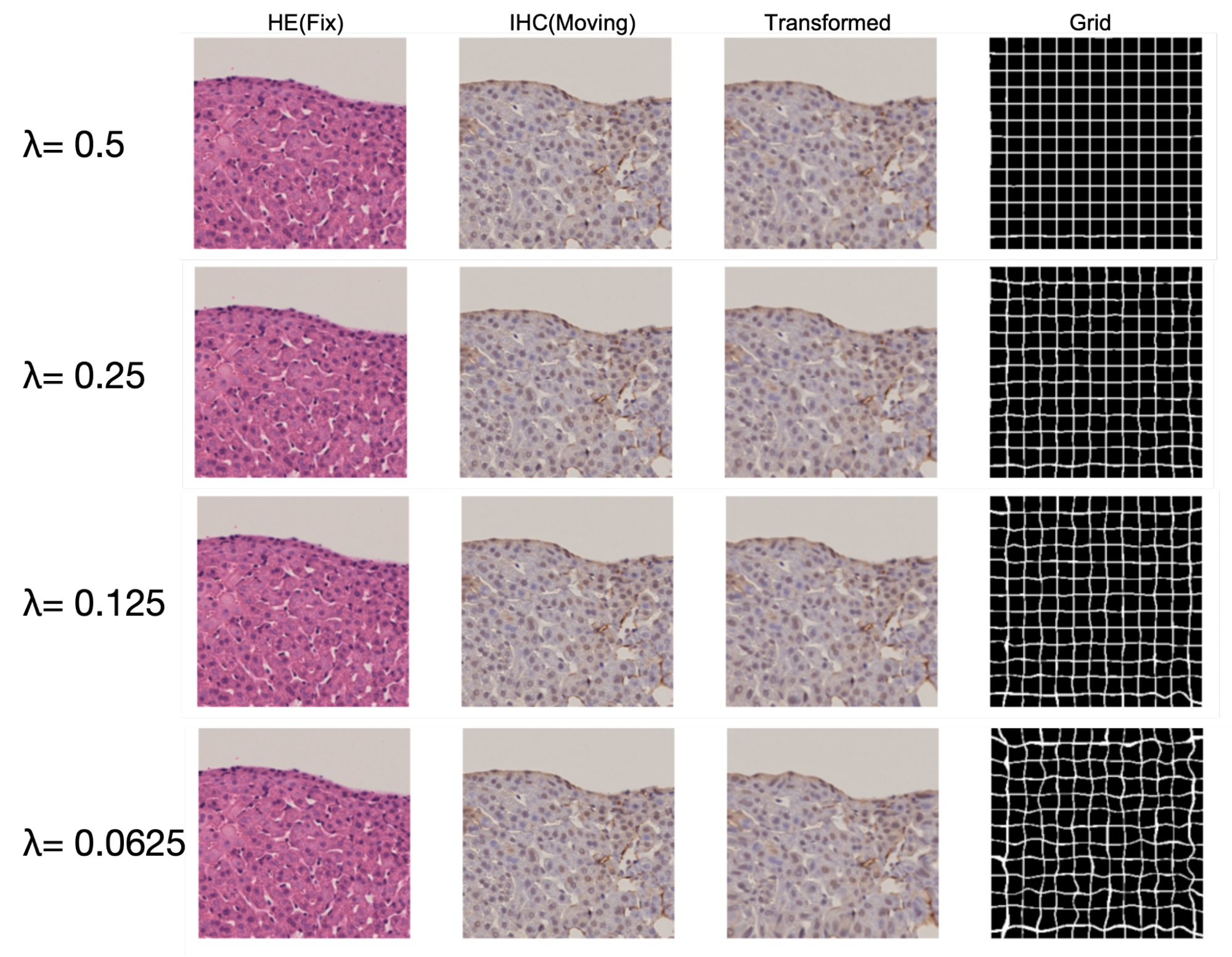
図9:正則化項を持つ各モデルのテストデータでの推論結果.
Discussion
今回の実験結果から,non-rigid registrationによるdeformableな変換で組織構造を歪ませる場合には,比較的強い正則化により変換ベクトル量を絞り込んで小さく抑えた方が,組織構造を保ちつつ,non-rigid registrationを行うのに適当であることがわかりました.
放射線画像データセットに対するVoxelMorphでは正則化項がなくても(\(\lambda\)=0),rigid registraiton後の画像に対して,non-rigid registrationである程度改善がみられたと報告されていますが[4],病理組織画像の場合は放射線科領域の画像よりも,より小さく複雑な構造(細胞,核など)が写っているので,正則化項がない場合だとdeformable fieldのパラメータの学習が適切に行えなかったのではないかと考えます.
今回作成したモデルはnon-rigid registationのみを行うモデルでした.しかしBackground部分でご説明したように,通常はrigid→ non-rigidの順でregistrationを行います.rigid registration部分で大まかに位置合わせを行います.しかし画像間の対象物体の位置のずれが大きくマッチングポイントの検出が不良な場合などでは,1回のrigid registrationで位置合わせがうまくいかないことがあります.そのような場合には,registrationを繰り返すことで,より正確な位置合わせを行なっていきます.今回のデータではprealignmentが比較的良好な画像が多かったのですが, 実際に病理診断で使われているWSIには, WSI間のずれが大きい, 標本に汚れが付着しているなどの理由で, prealignmentが不良で, non-rigid registrationを適用しても位置合わせ誤差の改善が望めない場合もあります. そのような場合, registration作業に占める貢献度はrigidの方がnon-rigidに比べて大きく,rigidとnon-rigidを一括して行い,registrationの作業回数を減らすように改良するとよりregistration用のモデルとして優れていると言えるのではないかと考えます.
また,WSIは,通常複数の倍率(対物レンズx5,x20,x40など)で撮像したデータがピラミッド型に積み重なったデータです.今回使用したデータは最高倍率(対物レンズx40)のデータのみを入力データとして使用していますが,低解像度の画像でrigid registrationを実施した後,その情報を保存しておき,高解像度の画像を用いてrigid + non-rigid registration行うことで,詳細な局所情報+大域的な情報を含んだより精度の高いregistration modelを作成できると考えられます.
Conclusion
今回の実験では,TransformerベースのAiRモデルのPatch embedding部分にCNNを導入し,損失関数に正則化項を加え, non-rigid registrationを行うモデルの開発を行いました.このモデルによるnon-rigid registrationでは,画像類似度や位置合わせ誤差の改善が認められました.
今後の課題としてregistration作業回数を減らすモデルやmulti resolutionのデータに対応したモデルの開発などが挙げられます.
Acknowledgements
本研究では東京大学大学院医学系研究科衛生学教室から提供されたWSIを使用し実験を行いました.貴重な試料を提供いただいた衛生学教室の皆様に心より感謝申し上げます.
最後に,約6週間という短い間でしたが,オンラインとオフライン(出社)のハイブリッドスタイルの中,社員の方のサポートのおかげで非常に充実したインターン生活を送ることできました.特にメンター,副メンター,アドバイザーの菅原さん,徳岡さん,岩澤さんには毎日のミーティングの中で色々な意見をいただき,多大なサポートをして頂きました.誠にありがとうございました.
References
- “ACROBAT 2022- Grand Challenge,” grand-challenge.org. https://acrobat.grand-challenge.org/Overview/ (accessed Sep. 29, 2022).
- Gatenbee, C. D., Baker, A. M., Prabhakaran, S., Slebos, R. J., Mandal, G., Mulholland, E., … & Anderson, A. R. (2021). VALIS: Virtual Alignment of pathoLogy Image Series. bioRxiv. https://doi.org/10.1101/2021.11.09.467917
- Wang, Z., & Delingette, H. (2021). Attention for image registration (air): an unsupervised transformer approach. arXiv preprint arXiv:2105.02282.https://doi.org/10.48550/arXiv.2105.02282
- Balakrishnan, G., Zhao, A., Sabuncu, M. R., Guttag, J., & Dalca, A. V. (2019). VoxelMorph: a learning framework for deformable medical image registration. IEEE transactions on medical imaging, 38(8), 1788-1800. https://doi.org/10.1109/TMI.2019.2897538
- Vos, B. D. D., Berendsen, F. F., Viergever, M. A., Staring, M., & Išgum, I. (2017). End-to-end unsupervised deformable image registration with a convolutional neural network. In Deep learning in medical image analysis and multimodal learning for clinical decision support (pp. 204-212). Springer, Cham. https://doi.org/10.1007/978-3-319-67558-9_24
- Marstal, K., Berendsen, F., Staring, M., & Klein, S. (2016). SimpleElastix: A user-friendly, multi-lingual library for medical image registration. In Proceedings of the IEEE conference on computer vision and pattern recognition workshops (pp. 134-142). https://doi.org/10.1109/CVPRW.2016.78.