Blog
本記事は、2023年夏季インターンシッププログラムで勤務された和田唯我さんによる寄稿です。
はじめに
PFN2023夏季インターンに参加させていただいた,慶應義塾大学理工学研究科修士1年の和田 唯我(Yuiga Wada)と申します.今回のインターンでは,今年4月にMeta社から公開された基盤モデルSegment Anything Model (SAM)を,三次元の医用画像へと適用するモデルの研究に取り組みました.本ブログでは,当研究の一部をご報告させて頂きます.
背景
Segment Anything Model (SAM) は,Meta社によって2023年4月に公開されたSegmentationタスクにおける基盤モデルです [1].本モデルは大規模言語モデル(LLM) に着想を得たモデルであり,LLMと同様,prompt(点, bounding box, 自然言語)を与えることができます.SAMは11億ものマスクを含む大規模データセットSA-1Bで学習されており,zero-shotでも高い性能を得られる点が最大の特徴です.

図1. SAMの生成するマスク例 [1]
SAMはその強力なzero-shot性能により,様々なドメインへの応用が期待されています.その中でも特に,アノテーション不足の問題等により大量のデータを確保することが難しい医用画像分野への応用が非常に有望視されています.その傍証として,SAMが公開された4月から今現在9月に至るまで,医用画像へSAMの適用を試みた先行研究が数多く行われています [2] [3] [4] [5] .
SAMの応用: 利点と課題
医用画像のデータセットは,患者様のプライバシーの問題や適切な知識を有した専門医がアノテーションする必要性など,様々な理由が相まって,学習に使えるデータ量が一般的なデータセットよりも少ない傾向にあります.したがって,医用画像を扱うモデルには,randomに初期化された重みから学習を始めるよりも,大規模なデータセットで事前学習された重みを初期値として用いたいというモチベーションが根強く,それゆえSAMのような基盤モデルを医用画像へ適用する研究が盛んに行われている次第になります.
しかし,SAMの3次元医用画像応用には次に示す3つの問題が存在します.すなわち, (i) 計算量 (ii) 3次元への拡張 (iii) prompt 依存性,の問題です.次節から,それぞれの課題について,本研究で行った実験を交えながら順に説明していきたいと思います.
実験結果
課題1. SAMの計算量
一つ目の課題はSAMの時間・空間計算量です.SAMは \(1024\times 1024\) の画像で学習されており,元論文 [1]によると,256枚の画像を学習するのに256GPUを使用しています.すなわち,1GPUに対して1枚の画像しか載らないほど,学習におけるメモリ消費が激しいということになります.そのため,SAMのfull-finetuningは現実的でなく,医用画像における研究のトレンドとして,decoderのみを再学習する手法 [2],ないしはAdapterやLoRA [6]のように学習可能パラメタを部分的に挿入するといった手法が採用される傾向にあります.
後述するように,医用画像は三次元であることが多いため,例えば各スライス方向(depth方向)に512枚画像が存在する場合\(1024 \times 1024 \times 512\) の画像をSAMに入力することになります.したがって,vanillaなSAMのままでは,上記内容からもわかるように,非現実的な時間・空間計算量を強いられることになります.そこで本研究では,SAMの性能を下げることなく時間・空間計算量を削減する方法を幾つか検討・調査しました.
SAMはViT [7] をベースに設計されているため,基本はTransformerベースです.したがって,画像の解像度を落としてもトークンの系列長が小さくなるだけで,意味のあるマスクが生成できるだろうという仮説を立て,実験的に \(512 \times 512\) の画像でSAMを推論(zero-shot)させました.図2にその結果を示します.実験には,腫瘍を含むCT医用画像で構成される Kidney Tumor Segmentation challenge 2021 dataset (KiTS21) [8] およびPancreas Tumor Segmentation task of 2018 MICCAI Medical Segmentation Decathlon challenge dataset (MSD-Pancreas) [9] を使用しました.
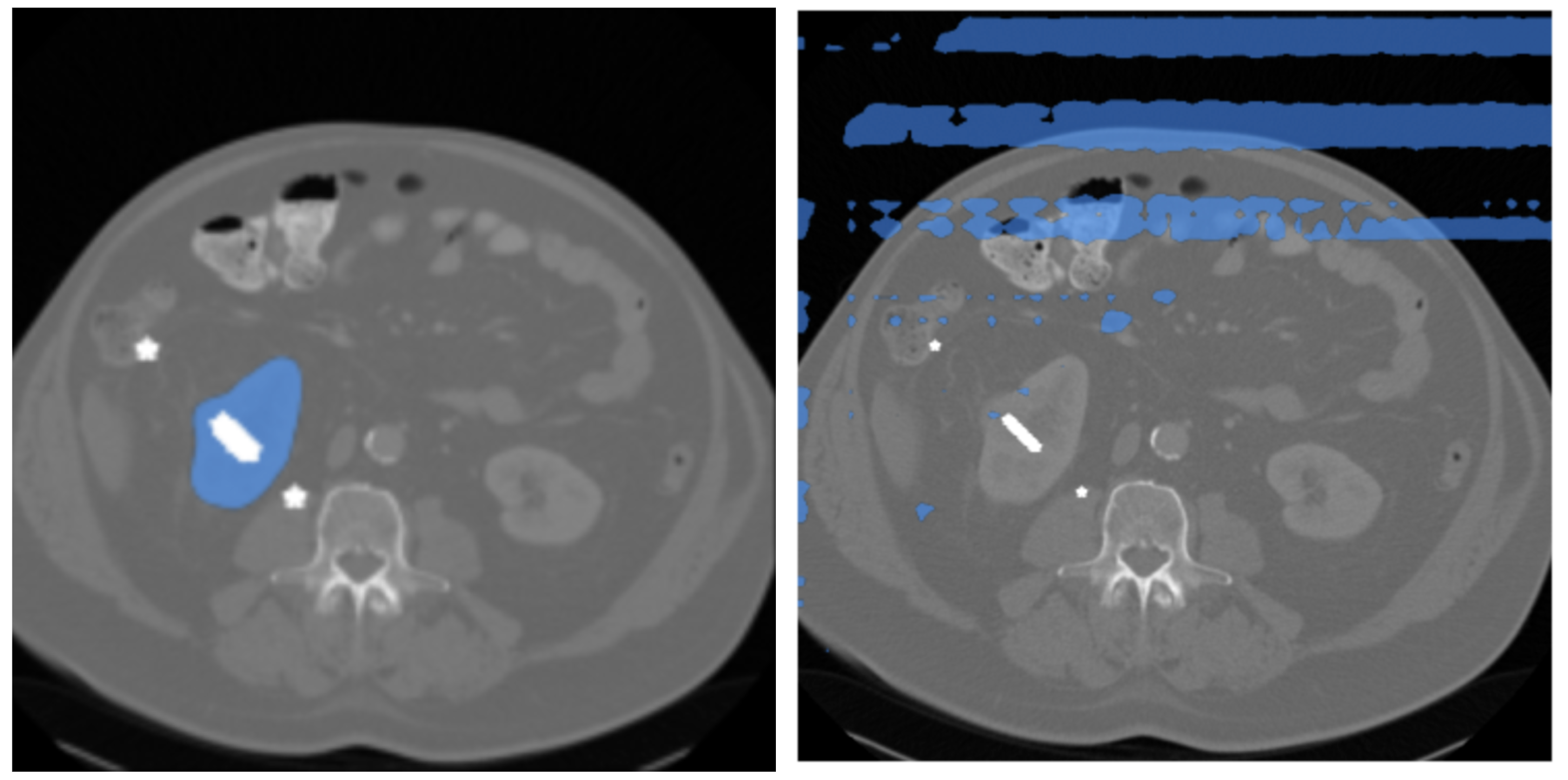
図2. SAM によるzero-shotの結果 (左: \(1024 \times 1024\) / 右: \(512 \times 512\))
図2を見ると,左の\(1024 \times 1024\)画像を入力した場合のマスクと比較して,右の\(512 \times 512\)画像では明らかに望ましくないマスクが得られていることがわかります.この結果から画像の解像度(系列長)に対してSAMはかなりsensitiveであることがわかりました.
上記実験はEncoder側の入力についての議論でしたので,次はEncoderではなくDecoder側の入力の系列長を調整し,その挙動を調査する実験を行いました.具体的には,図3に示すようにトークンを所望の系列長になるようpoolingし,その後interpolateして元のサイズに復元しています.
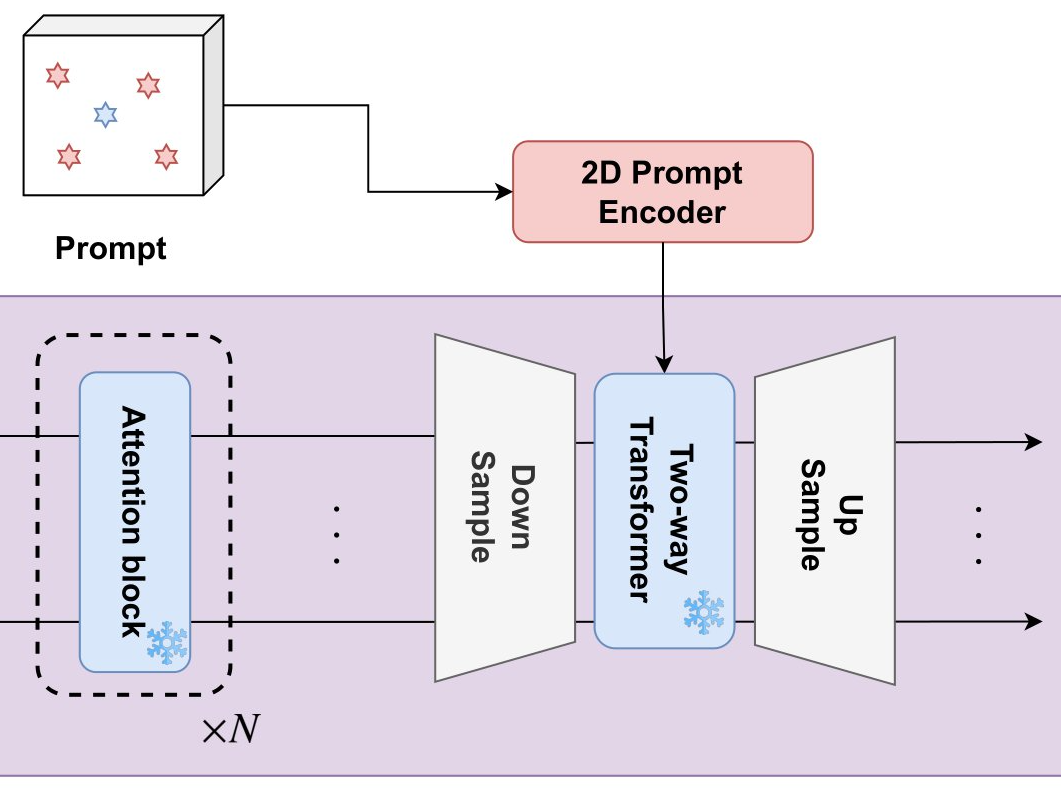
図3. Decoderでの系列長の調整に関する概略図
図4にその結果を示します.
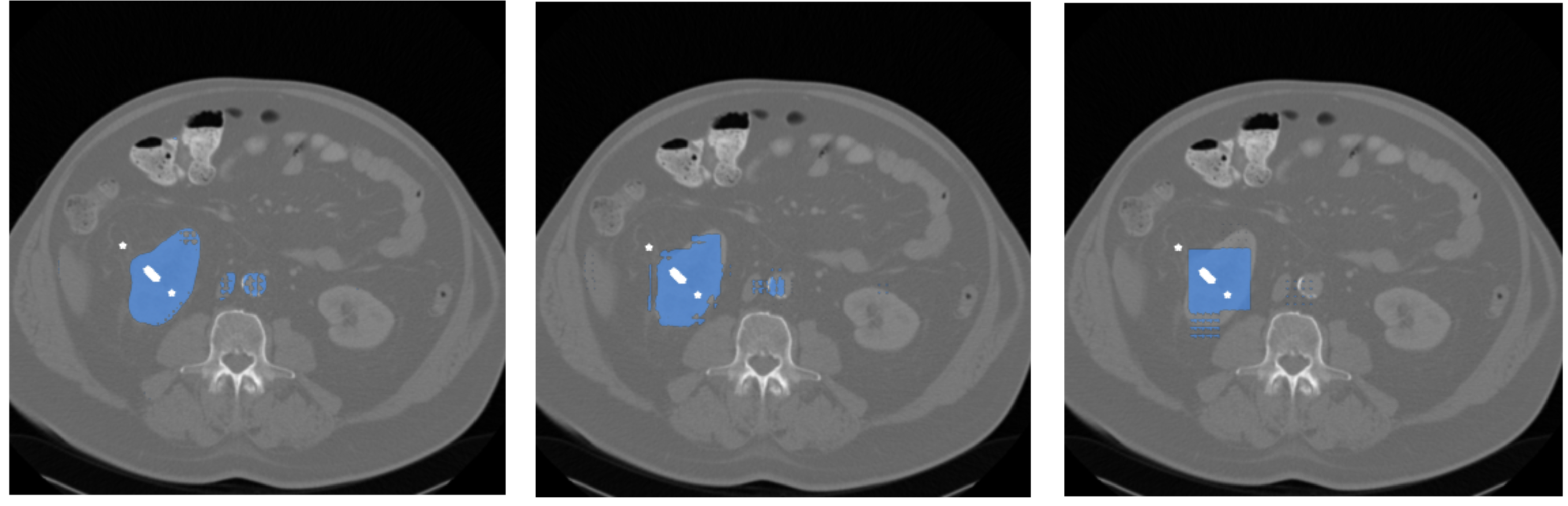
図4. 左: 系列長の調整なし / 中央: 系列長を1/2に調整 / 右: 系列長を1/4に調整 (白点は prompt を示しています)
図4を見てみると,Encoderでは入力の系列長にsensitiveであったのに対して,Decoderでは系列長に対する制約が緩いことが分かりました.
これらの実験により,三次元医用画像にSAMを適用する上で課題となっていた計算量の問題に関して,SAMのもつ特性やSAMの計算量削減の知見を得ることが出来ました.SAMの計算量に関しては,皆同じ課題感を持っているようで,最近はMobile SAM [11] やFast SAM [12]といった高速に動作しうるSAMの代替モデルが登場してきています.
課題2. SAMの三次元への拡張
二つ目の問題は「SAMを如何に三次元へと拡張するか」という問題です.SAMを三次元画像に適用した代表的な先行研究として,3DSAM-adapter [4] とMedical SAM Adapter (MSA) [5] があります.本研究では両者共に動作確認を行いましたが,残念ながら後者のMSAでは正しい動作が得られず,論文値を再現することができませんでした.したがって,本インターンでは,主に3DSAM-adapterに焦点を当てて研究を進めていきました.
3DSAM-adapter: モデル構造
図5に3DSAM-adapterのモデル構造を示します.
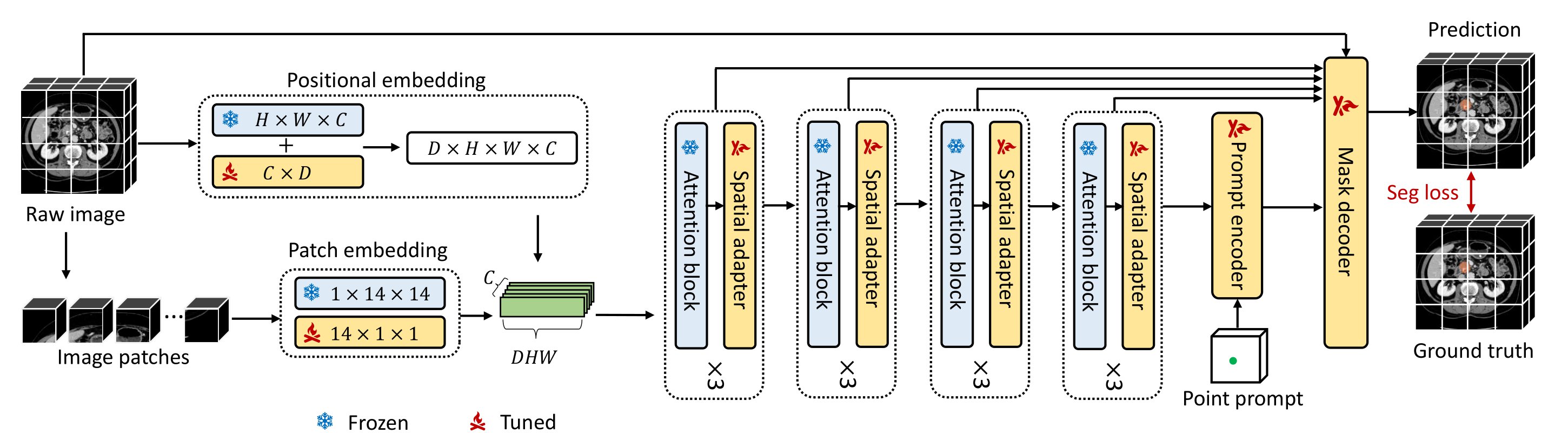
図5. 3DSAM-adapterのモデル図 [4]
3DSAM-adapterは3次元医用画像(以下 3D volume)を2Dとして処理するのではなくそのまま3Dとして処理することができます.このとき,本モデルではSAMの重みを最大限活用するため,EncoderはAdapter型の拡張がなされており,Adapter以外は重みが固定される形で学習されます.具体的には,橙色で表されているAdapterが各ブロックに挿入され,EncoderにおいてはAdapterのみが学習される形となります.ただし,3D volumeを扱う都合上,Prompt EncoderとDecoderは元のSAMとはやや異なる構造を有しているためSAMの重みを引き継いでおらず,またEncoderとは異なり全パラメタが学習対象となっています.
図6にAdapterのアーキテクチャを示します.
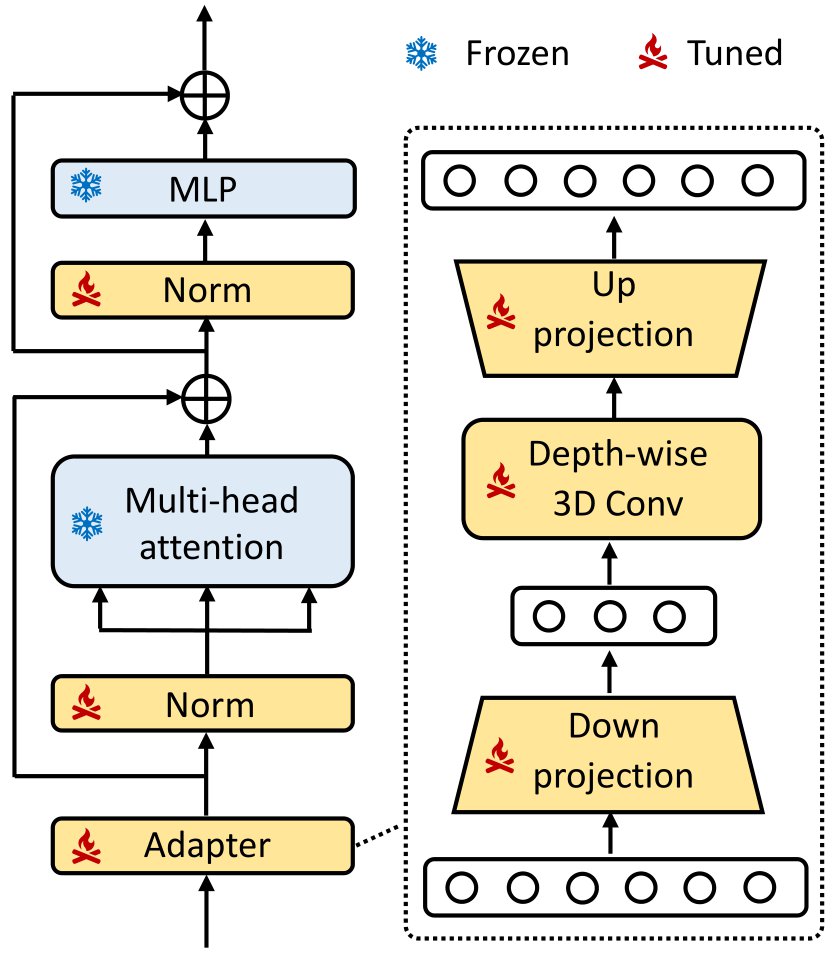
図6. Adapterのアーキテクチャ [4]
Adapterは以下の形で定式化されます. \[\text{Adapter}(\textbf{X}) = \textbf{X}+\sigma(\textbf{X}W_{down})W_{up}\]
ここで, \(\textbf{X} \in \mathbb{R}^{N\times c}\) は入力となる特徴量, \(W_{down} \in \mathbb{R}^{c\times m}\) および \(W_{up} \in \mathbb{R}^{m\times c}\) はそれぞれDown-Projection, Up-Projectionの重みを指し,\(\sigma(\cdot)\) は活性化関数を表します.このように,3DSAM-adapterではbottleneck型のAdapterを採用し,軽量なAdapterを実現しています.
3DSAM-adapterは3D volumeを3Dとして扱うため,様々な工夫が成されています.例えば,通常のSAMでは\(1 \times 16 \times 16\)のPatch Embeddingが行われる一方で,本モデルでは更に\(16 \times 1 \times 1\)のPatch Embeddingを行い,\(16 \times 16 \times 16\)の3D Convolutionに近似されうるような3次元Patch Embeddingを実現しています.さらに本モデルでは,\(D \times H \times W\) の3D volumeを扱う上で,スライス方向にflattenし,通常のSAMへ入力できる形に変形するのですが,前述の計算量の問題を避けるために,Swin Transformer [10] のアイデアを参考にWindow Attentionを適用することで計算量を削減しています.
具体的な入出力の例を図7に示します.実験ではA100を4台使って3DSAM-adapterを学習させています.
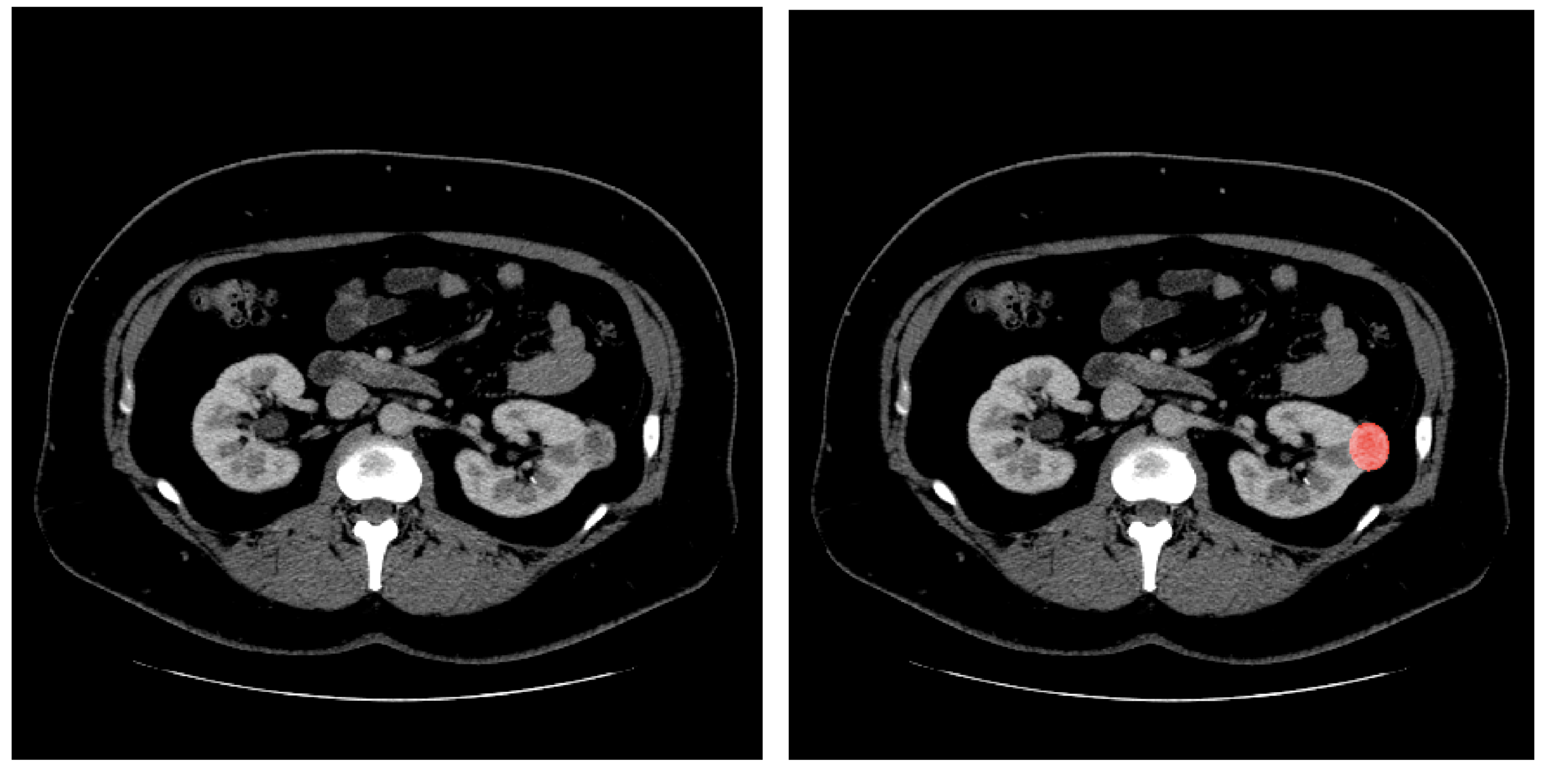
図7. 3DSAM-adapterにおける入出力の例 (左: 入力, 右: 出力)
3DSAM-adapter: 問題設定
次に,3DSAM-adapterにおける問題設定について説明します.通常のSAMは \(1024 \times 1024\) 画像を入力として学習しますが,3DSAM-adapterでは全体volumeから腫瘍を含むようcropしたsub-volume(例えば\(128 \times 128\))を\(512 \times 512 \times 512\) へと拡大したものを入力とします.(図8に例を示します)
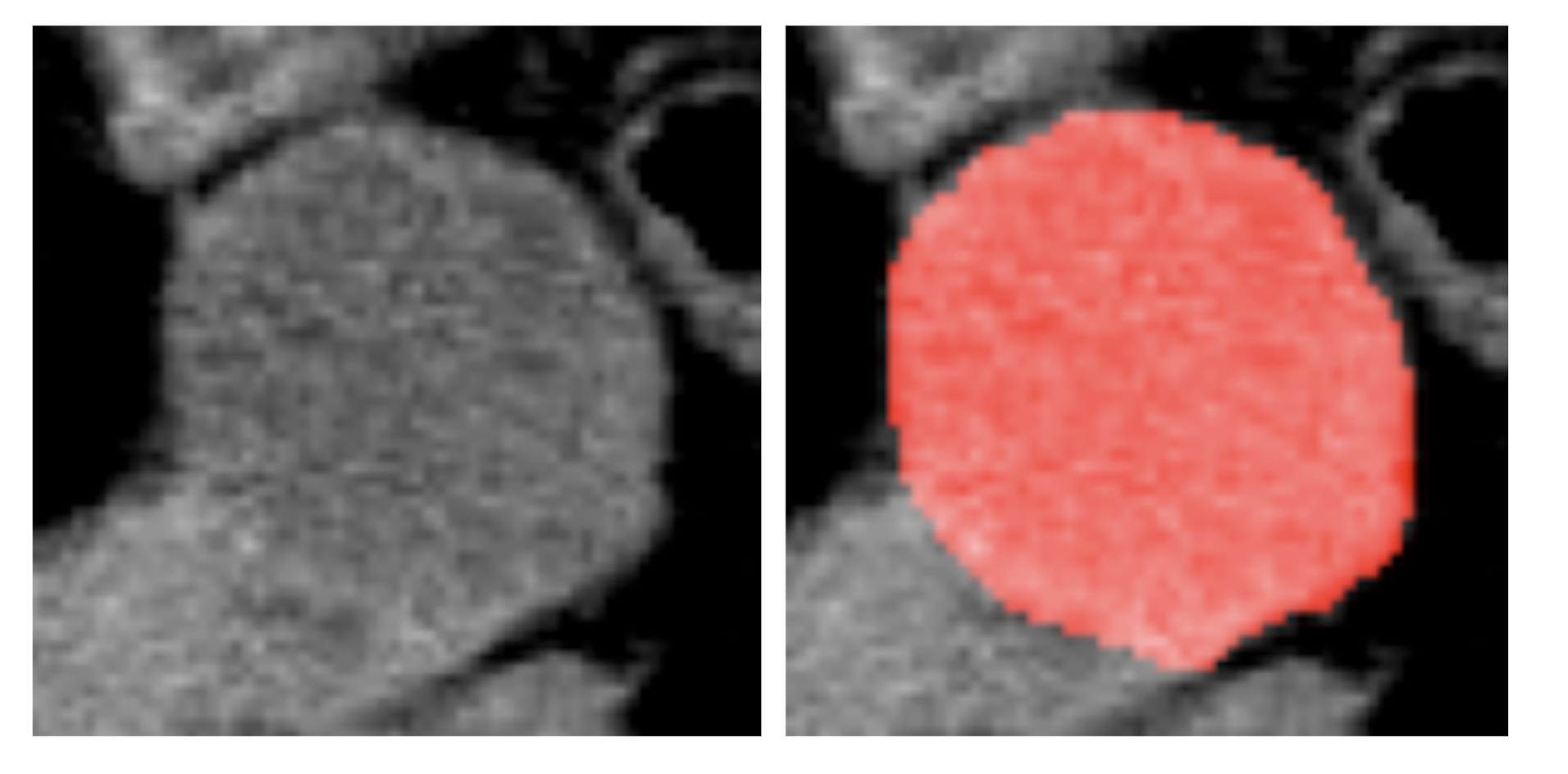
図8. 左: 実際にcropされた画像 / 右: マスクGround Truth (GT)
そのため,whole-volumeを扱う際よりも腫瘍が相対的に大きくなることでタスクの難易度が易化している可能性や,大域的な情報が欠けていることにより過度に偽陽性が生じる可能性があります.
このとき,上記のような問題設定では,我々が課題1の実験で示した通り,3DSAM-adapterにおけるEncoderの入力が,本来のSAMの事前分布から外れたものとなっている可能性があります.さらに,前述の通りPrompt EncoderとDecoderはSAMの重みを使わずrandom initizalizeされた重みを使用しているため,promptを正しく理解しているかどうかも詳しく精査する必要があります.そのため,本研究ではこれらの観点を再度考察し直すためにも追加実験を行いました.
3DSAM-adapterの追加実験
前節では,3DSAM-adapterが3D volumeをcropしているためにタスクが易化している可能性について言及しました.そこで,本研究ではcrop sizeによる3DSAM-adapterの性能差を調査しました.表1にその結果を示します.ただし,元論文では,腫瘍を中心としてcropした画像に対してテスト評価を行っているため,表1の評価も同様の評価法に準じていることに留意してください.
| Crop size | Original [%] |
| 128×128 | 75.9 |
| 256×256 | 54.9 |
| 512×512 | 32.3 |
表1を見てみると,確かにcrop sizeが大きくなるにつれて,タスクのDiceスコアが低下していることが見て取れます.このことから,確かに問題設定によって腫瘍のsegmentationタスクそのものが易化している可能性が見て取れます.
次に,crop sizeが \(128 \times 128\)である設定で,sliding window inferenceによる推論を行った際の定性結果を図9に示します.
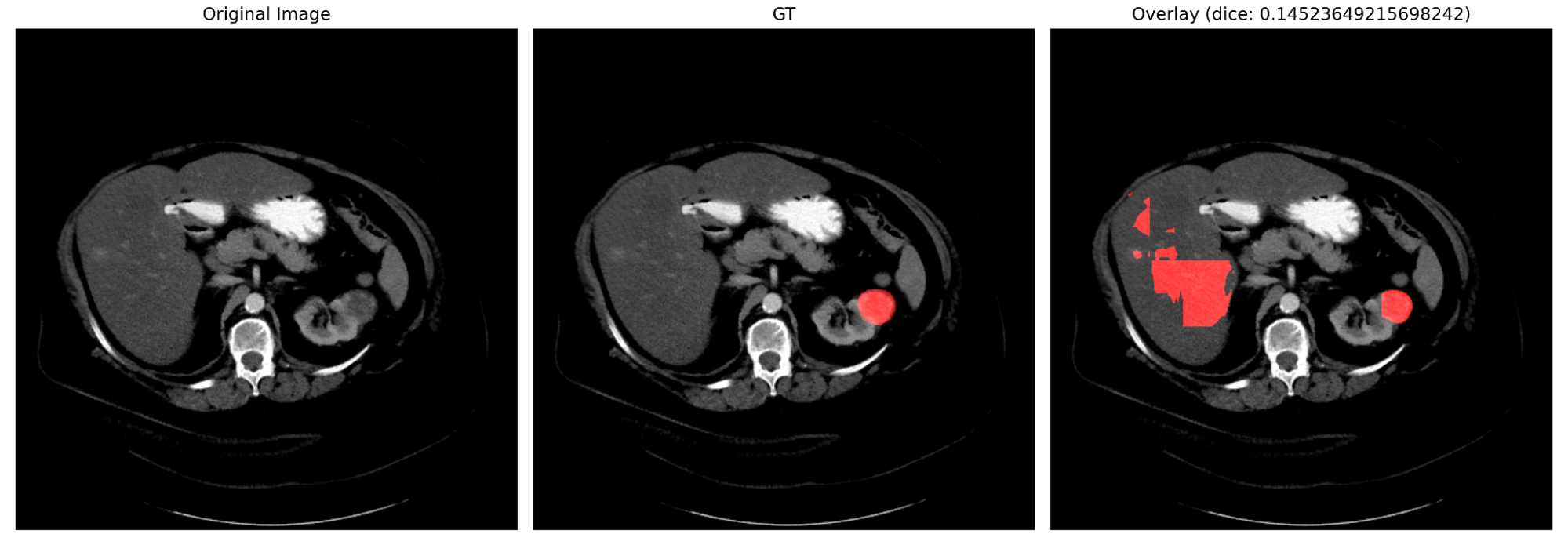
図9. Sliding window inferenceによる推論結果(左: GT, 右: 推論結果)
上図を見てみると,腫瘍のマスクをある程度正確に予測できている一方で,かなり歪な偽陽性が生じていることがわかります.
前節では,Prompt EncoderとDecoderはSAMの重みを使わずrandom initizalizeされた重みを使用している旨を記述しました.そこで,本研究では,3DSAM-adapterが適切にpromptを捉えているかを調査する実験も行いました.
表2は,promptを適切に与えた場合 (Original),ランダムに与えた場合 (Random prompts),およびPrompt Encoderをモデルから取り除いた場合 (w/o Prompt-Encoder) の三条件で実験した結果を示しています.
| Crop size | Original [%] | Random prompts [%] | w/o Prompt-Encoder [%] |
| 128×128 | 75.9 | 77.3 | 74.2 |
| 256×256 | 54.9 | 54.0 | 53.9 |
| 512×512 | 32.3 | 32.3 | 32.3 |
表2を見てみると,promptをランダムに与えても,通常の3DSAM-adapterとほぼ性能が変わらないという結果が得られました.更に,Prompt Encoder自体を取り除いたとしても,性能差がないという結果も得ることができました.
この結果より,やはりPrompt EncoderとDecoderがSAMの重みを引き継いでいないことから,promptを適切に捉えきれていない可能性が示唆されます.
このことを更に精査するため,定性結果も見てみます.図10に,腫瘍を指すpositive promptを過剰に与えた場合の定性結果を示します.また図11に,腫瘍以外をpositive promptとして与えた場合の定性結果を示します.
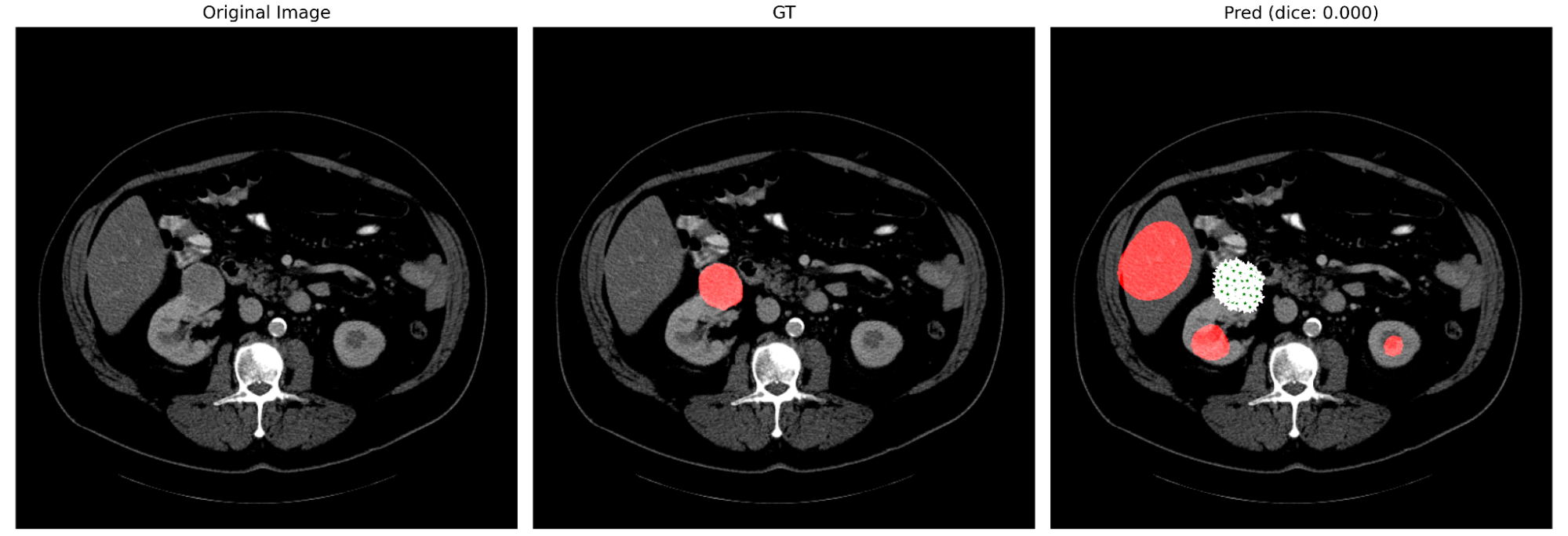
図10. positive promptを過剰に与えた場合 (prompt は緑の星で示されています)
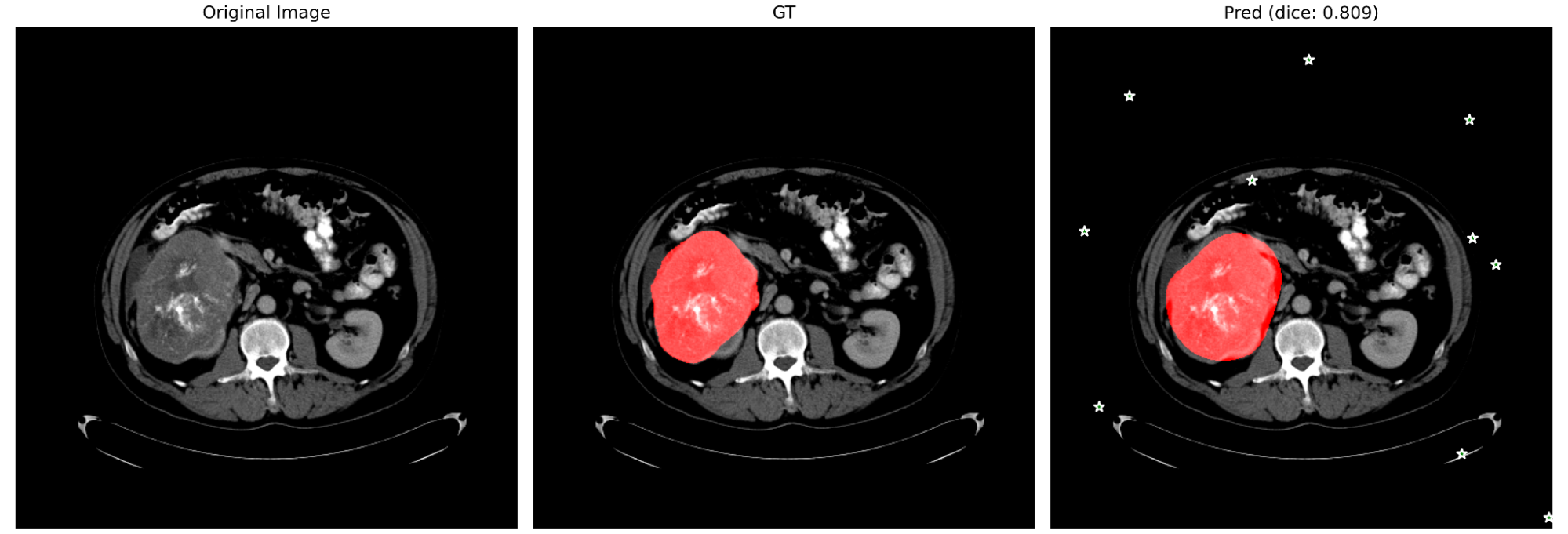
図11. 腫瘍以外をpositive promptとして与えた場合 (prompt は緑の星で示されています)
図10, 11より,promptをどれだけ適切に与えても,また逆にどれだけ無作為に与えても,promptを無視して予測している様子が見て取れます.これらの結果からも,やはり3DSAM-adapterがpromptを適切に捉えきれていない可能性が示唆されます.
課題3. SAMのprompt依存性
三つ目の問題はSAMの「強いprompt依存性」です.(注: 課題2で実験した 3DSAM adapter では Prompt Encoder および Mask Decoder を一から学習しているため,表2のように prompt 依存性が全く無いように見えます.しかし,vanilla SAM の Mask Decoder 等を使う際には依然 promp 依存性が問題となります.)SAMは前述の通りLLMに着想を得たモデルであるため,promptを与えることを前提としたモデル設計が成されています.そのため,医用画像のautomatic segmentationを志向する研究は少なく[3],むしろこのSAMの特性を生かしたinteractive segmentationとして問題を設定する研究がマジョリティになっています [2][4][5].ここで,interactive segmentationとは,ユーザがある3次元点群ないしはbounding boxをプロットした際に,その点をガイドとしてターゲット(腫瘍ないしは臓器)のマスクを生成する類のsegmentationタスクを指します.今回の文脈で言えば,ターゲットの三次元点群を予め数点だけモデルに与え,そのターゲットの正確なマスクを生成させるといったタスクになります.
しかし, 見落としの防止が重要な価値となる医用画像解析において,本来我々が欲しているのは,ユーザが腫瘍の位置を事前に把握しているinteractiveな手法ではなく,腫瘍の位置が分からない状況で,モデルが自動的に腫瘍のmaskを生成してくれるautomaticな手法です.そのため,こうしてautomaticにmaskを生成したいという目的においては,腫瘍の位置を予め与えておくinteractive segmentationの問題設定はあまり好ましいとは言えません.本研究では,SAMのprompt依存性に関する実験も実施しましたが,ここでは詳細を割愛させていただきます.
結論
今回,SAMを三次元医用画像へ適用する際の利点と課題について,また3DSAM-adapterの問題点について,様々な実験を通してご紹介しました.本研究で行った諸実験により,SAMを医用画像へ適用する際には,SAMの事前分布に可能なだけ準じる必要があること,またその制約を満たすには計算量がボトルネックになってくるという二点が示唆されました.
今後の課題としては,これら二点の考察から,SAMの事前分布に準じるよう各スライスを独立に処理する機構や,三次元方向に特徴量をmixする機構の開発などが考えられます.
おわりに
最後に,岩澤さん,菅原さん,徳岡さんをはじめとするPFN社員の皆様,およびその他ご協力いただいた皆様にこの場を借りて御礼申し上げます.2ヶ月弱という短い期間でしたが,貴重な機会をいただきまして,誠に有難うございました.
参考文献
- Kirillov, Alexander, et al. “Segment anything.” arXiv preprint arXiv:2304.02643 (2023).
- Ma, Jun, and Bo Wang. “Segment anything in medical images.” arXiv preprint arXiv:2304.12306 (2023).
- Gao, Yifan, et al. “DeSAM: Decoupling Segment Anything Model for Generalizable Medical Image Segmentation.” arXiv preprint arXiv:2306.00499 (2023).
- Gong, Shizhan, et al. “3DSAM-adapter: Holistic Adaptation of SAM from 2D to 3D for Promptable Medical Image Segmentation.” arXiv preprint arXiv:2306.13465 (2023).
- Wu, Junde, et al. “Medical sam adapter: Adapting segment anything model for medical image segmentation.” arXiv preprint arXiv:2304.12620 (2023).
- Hu, Edward J., et al. “LoRA: Low-Rank Adaptation of Large Language Models.” ICLR. 2021.
- Dosovitskiy, Alexey, et al. “An image is worth 16×16 words: Transformers for image recognition at scale.” ICLR. 2021.
- Heller, Nicholas, et al. “The state of the art in kidney and kidney tumor segmentation in contrast-enhanced CT imaging: Results of the KiTS19 challenge.” Medical image analysis. 2021
- Michela Antonelli, et al. “The medical segmentation decathlon”. Nature communications 13(1), 4128. 2022.
- Liu, Ze, et al. “Swin transformer: Hierarchical vision transformer using shifted windows.” ICCV. 2021
- Zhang, Chaoning, et al. “Faster Segment Anything: Towards Lightweight SAM for Mobile Applications.” arXiv preprint arXiv:2306.14289 (2023).
- Zhao, Xu, et al. “Fast Segment Anything.” arXiv preprint arXiv:2306.12156 (2023).