Blog
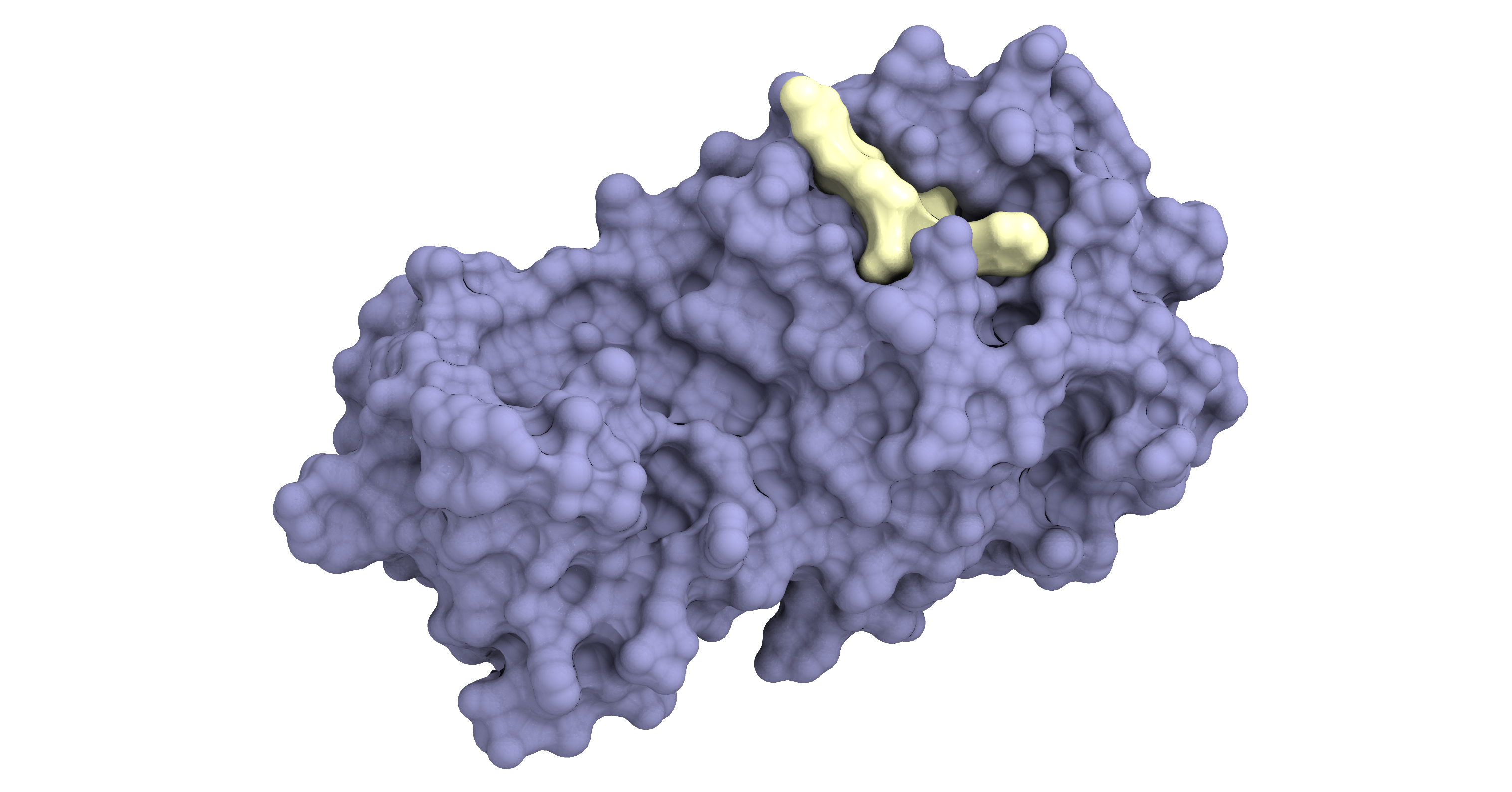
新型コロナウイルスのメインプロテアーゼ (紫色)に結合して増殖を阻害する薬剤 (黄色)
はじめに
新型コロナウイルス感染症 (COVID-19) は、現在、ワクチンの開発と接種により、感染の拡大が抑えられることが期待されているものの、ウイルスのさらなる変異によるパンデミックの再発を防止するには、SARS-CoV-2を標的とした治療薬の開発が強く望まれています。
レムデシビルなどの核酸合成系を標的とする化合物やREGN-COV2のようなウイルスの抗体を直接注入する治療法は一定の臨床効果が報告され認可されていますが、限定的な用途に限られています。それ以外にも、多くの既存薬剤についてドラッグリポジショニングの研究が行われていますが、認可を得るまでには至っていません。また、変異ウイルスへの脅威に対応するためにも、多様なCOVID-19の治療薬の開発が望まれています。
3CLプロテアーゼ (Mpro) はウイルスが増殖するためには必須のタンパク質であるとともに、ヒトにはホモロジーが高い酵素は認められていないため、ウイルス病治療薬の標的として有力と考えられています。一例として、ライノウイルスのMpro阻害剤として Rupintrivirが知られていますが、COVID-19治療薬としては実用化に至っていません。また、これまでに知られているMpro阻害剤はロイシンあるいはその類似の構造を持つペプチド様化合物に集中していますが、将来のウイルスの変異に備えるためにも異なる構造をもつリード化合物の発見が望まれます。
PFNは、SARSウイルスやMERSウイルスといったコロナウイルスのMpro阻害剤の探索研究を長年行ってきた京都薬科大学の赤路健一教授、小林数也准教授との共同研究により、COVID-19の治療薬候補となるこれまでと異なる構造を持つMpro阻害剤を見出しました。
PFNのAI創薬プラットフォーム
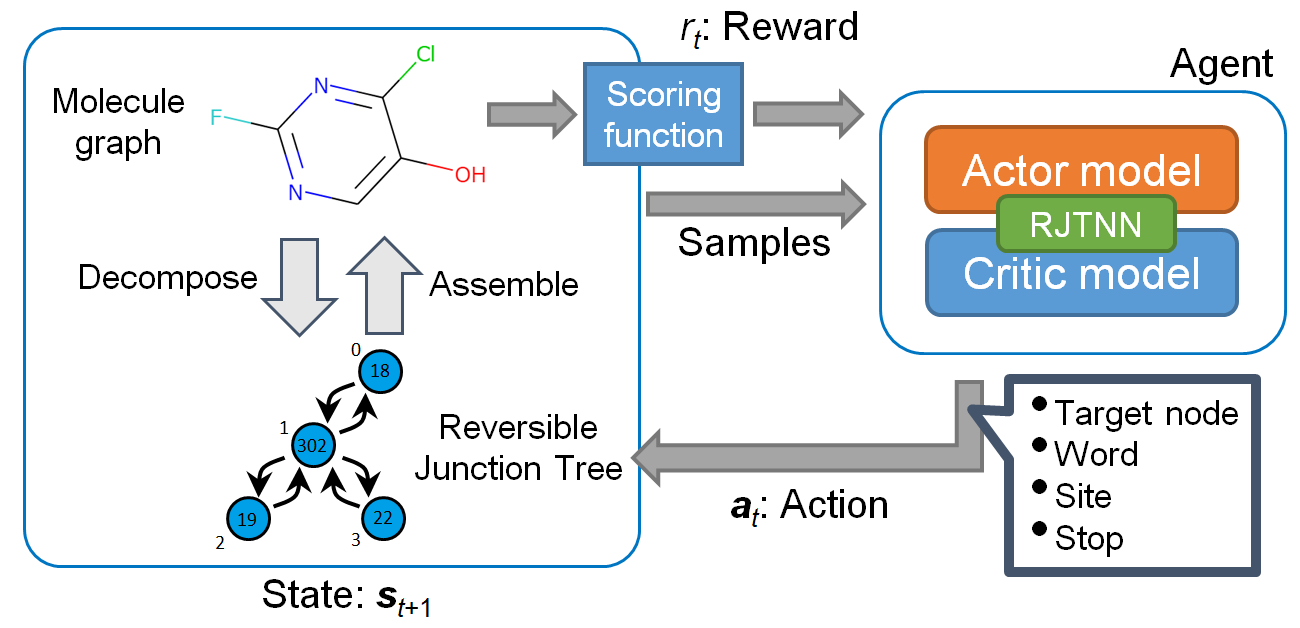
近年、機械学習による画像の識別や生成は目覚ましい発展をしていますが、この方法を化合物デザインへ応用する研究 (いわゆるAI創薬) が注目を集めています。PFNでは深層学習と豊富な計算能力を武器に、独自のAI創薬プラットフォームを構築しています。本プラットフォームは、人手やルールベースによる構造修飾により設計された化合物や化合物ライブラリによるバーチャルスクリーニングを実施するだけでなく、種々の化合物生成モデルとOptunaなどに実装されている最適化アルゴリズムを組み合わせることで化合物の設計と最適化を迅速に行い、化合物ライブラリに含まれない構造や、従来の方法では着想しにくい構造の提案も可能としています (図1)。
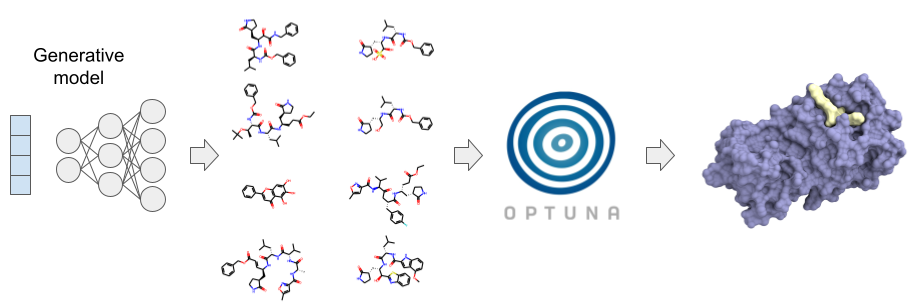
図1: PFNの創薬プラットフォームの概念図.
SARS-CoV-2 Mpro阻害剤の設計
これまでの赤路教授の研究により、図2に示すペプチド性化合物 (左) や非ペプチド化合物 (右) が、SARSウイルスのMproを阻害することが明らかとなっています[1]。 今回の共同研究では、PFNのプラットフォームを活用して、AIによる仮想空間での分子設計と分子モデリングを実施し、SARS-CoV-2のMproを阻害する、より単純かつ非ペプチド様の化合物の発見を目指しました。提案された化合物から13化合物を現実世界で合成、活性試験を実施した結果、7化合物がSARS-CoV-2 のMproに対する阻害活性を示し、新型コロナウイルス治療薬探索のリード化合物として有力であることがわかりました。このリード化合物を治療薬としていくためには、今後も多くの研究を積み重ねていく必要がありますが、本研究結果はPFNのAI創薬プラットフォームが現実世界の創薬研究に応用可能であることを示すものであると考えています。
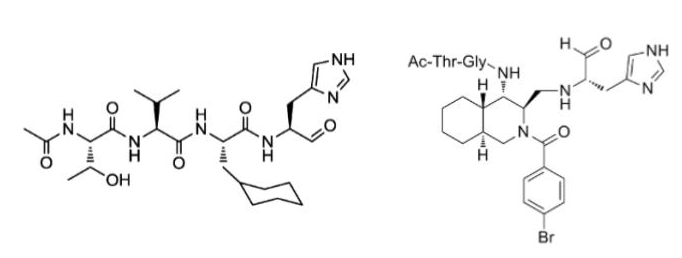
図2 ペプチド性化合物 (左, 文献[2])および非ペプチド性化合物 (右, 文献[3])の構造.
おわりに
本研究で得られた成果は、COVID-19治療薬の開発に新しいアプローチができたとともに、カテプシンやカスパーゼといったシステインプロテアーゼを標的とする創薬や、ペプチド様リガンドからペプチド構造脱却を指向したスキャフォールドホッピングへの応用も可能であると考えています。PFNは今後もAI創薬技術の改良を続けるとともに、AI創薬に共同で取り組むパートナーを募集し、実用化に向けた研究を加速させていきます。
参考文献
[1] Akaji, K. YAKUGAKU ZASSHI 2021, 141, 215-233.
[2] Akaji, K. et al. J. Med. Chem. 2011, 54, 7962-7973.
[3] Ohnishi, K. et al. Bioorg. Med. Chem. 2019, 27, 425-435.
Tag