Blog
本記事は、2023年夏季インターンシッププログラムで勤務された菱沼秀和さんによる寄稿です。
Introduction
2023年度夏季インターンシップに参加した九州大学大学院 医学専攻 博士課程3年の菱沼 秀和と申します。今回のインターンシップでは、画像セグメンテーションの基盤モデルである Segment Anything Model (SAM) の医用画像に対する応用手法について研究しました。インターンシップ中の取り組みのうち、本記事では特に SAM とその派生モデルの比較や fine-tuning の手法についての結果を紹介します。
Background
SAM [1] は2023年4月に Meta 社が発表した画像セグメンテーションのための基盤モデルです。約1100万枚の画像と10億以上のセグメンテーションマスクから学習されており、追加で学習を必要としない zero-shot により優れたセグメンテーションを行える点が注目されています。SAM の重要な特徴として、何をセグメンテーションしたいかについての情報をプロンプトとして与える事で目的のマスクが得られやすいという性質があります。プロンプトは具体的には、関心のある領域の位置を示す点や bounding box、大雑把なマスクなどの空間情報、テキスト形式などの意味的情報として与えられます。ただし、論文中ではテキストプロンプトに対応していると記載されているものの、2023年9月現在テキストプロンプトを入力する機構の実装は公開されていません。
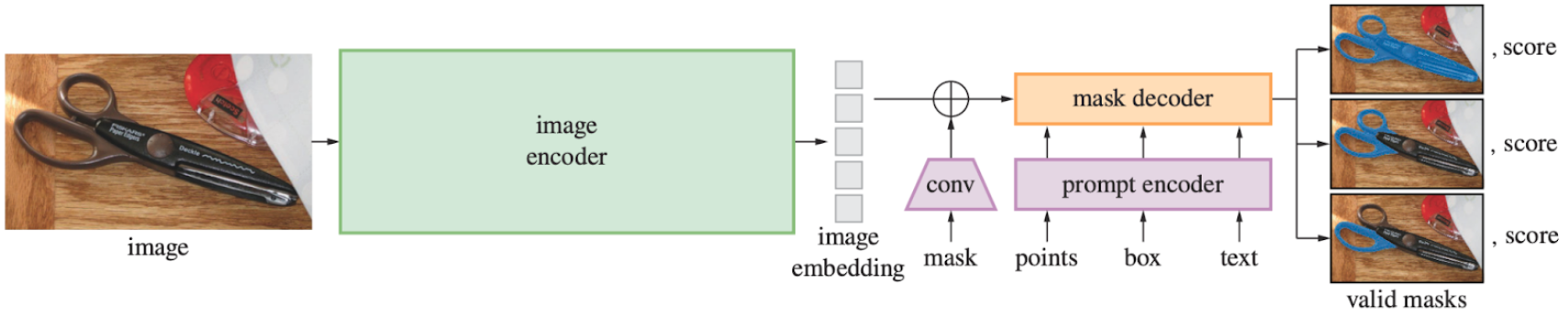
図1. Segment Anything Model (SAM) の概略図 (Kirillov et al. 2023 [1] Fig.4より引用)
SAM は大きく分けて、Image Encoder, Prompt Encoder, Mask Decoder の3つの構造からなります (図1)。セグメンテーションしたい画像の入力は Image Encoder を通してベクトルに embedding されます。プロンプトは Prompt Encoder に入力し embedding され、この二つが Mask Decoder 内で統合されマスクが出力されます。
SAM は様々な画像に対して高い精度が得られる一方、医用画像のセグメンテーションに関する精度は十分とは言えません。なぜなら SAM の訓練データには医用画像が含まれていないからです。医用画像と SAM の訓練に用いられた自然画像には大きな domain gap があるため、SAM を活用して zero-shot で医用画像のセグメンテーションを行うことは困難だと考えられます。SAM が発表されてからこのインターンシップが始まるまでの四ヶ月の間にも、SAM を医用画像に適用するための手法が提案されてきました。そのような先行研究のうち、今回のインターンシップでは MedSAM と AdaptiveSAM と呼ばれる2つのモデルについて着目しました。
MedSAM [2] は CT や MRI、病理画像などの計100万枚以上にのぼる様々な医用画像で構成されたデータセットを用いて SAM を fine-tuning したモデルです。SAM と比較して、MedSAM は細かい形状の臓器や病変をより正確にセグメンテーション可能であることを示しています。
AdaptiveSAM [3] は手術動画のスナップショットからテキストで指定した臓器や器具のマスクを予測するように SAM を改良したモデルです。AdaptiveSAM の特徴は医用画像のためのSAMを元にしたモデルのうち、著者の知る限り唯一テキスト形式のプロンプトに対応できるように拡張している点にあります。加えて、小規模なデータセットで Transformer を fine-tuning する際にはバイアス項の変更のみで効率よく学習が行えるという先行研究 [4] の知見を取り入れていることから、AdaptiveSAM は学習可能なパラメータが特徴的な組み合わせとなっています。
現在、医用画像セグメンテーションのための SAM の fine-tuning 手法について、デファクトスタンダードなどは定まっていません。そこで今回私たちは医用画像セグメンテーションに最も効果的な fine-tuning 手法を探るために、bias-tuning を含む複数の fine-tuning の手法を比較しました。
Results
Federated Tumor Segmentation (FeTS) Challenge 2022 dataset
Fine-tuning 手法の比較にあたり、データセットは Federated Tumor Segmentation (FeTS) Challenge 2022 [5] で提供されたものを用いました。このデータセットは23の施設から脳腫瘍を持つ患者の脳 MRI を収集したもので、合計1,251症例が含まれます。
MRI の画像には、図2のようにT1強調、造影剤投与後のT1強調、T2強調、T2-FLAIR の4チャンネルが含まれます。脳腫瘍のアノテーションは図3のように腫瘍をさらに GD-enhancing tumor (ET), peritumoral edematous/invaded tissue (ED), necrotic tumor core (NCR) の3種類のラベルに分類してアノテーションされています。今回の fine-tuning 手法の比較では、画像の入力は造影剤投与後T1強調の1チャンネルのみで、ET 領域のセグメンテーションをタスクとしました。ET 領域は典型的には造影剤を用いた際にリング状に映る領域です。
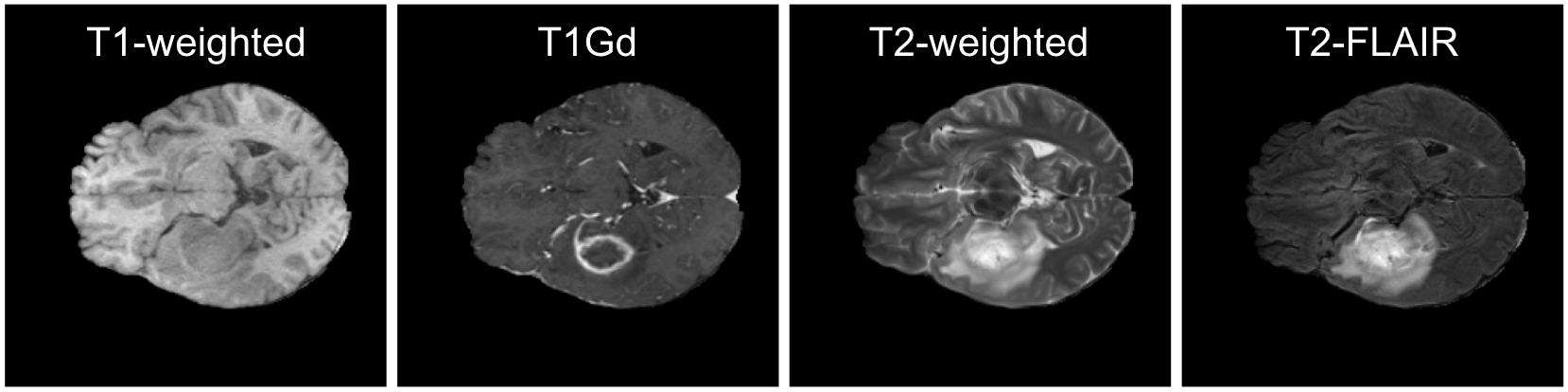
図2. 脳 MRI の4種のモダリティの例 (FeTS2022 [5] より作成)
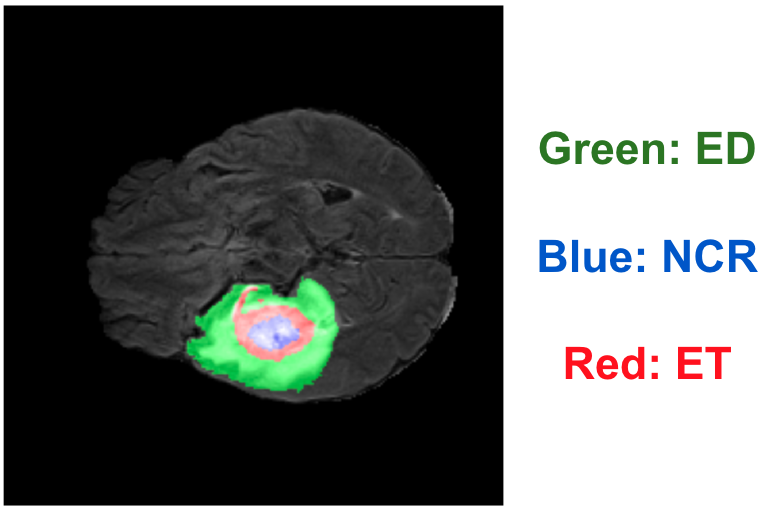
図3. 脳 MRI の3種のアノテーションの例 (FeTS2022 [5] より作成)
脳MRIを提供した施設ごとの症例数には大きくばらつきがあります。今回、10未満の症例を持つ14施設については training データと validation データから除外して test データとし、10以上の症例を持つ施設は1割を test データとしました。すなわち test データは、学習したデータと同じ施設 (In-Distribution) と、1つのサンプルも学習されていない施設 (Out-Of-Distribution; OOD) の両方の症例画像から構成されます。
評価指標には、セグメンテーションタスクで一般的に用いられる Dice スコアを用いました。Dice スコアは予測したセグメンテーション領域と正解領域の重なりが大きいほど高い値として評価できる指標です。
Model の比較
Fine-tuning の手法を検討するにあたり、事前に FeTS2022 データセットを用いた腫瘍全ラベルのセグメンテーションタスクにおいて fine-tuning を行っていない SAM と MedSAM の差を比較しました。各モデルの出力の例を図4に示します。プロンプトとして与えた bounding box に基づいて、SAM も MedSAM もセグメンテーションを行っていますが、MedSAM の方が腫瘍と正常組織の境界をより正確に捉えています。セグメンテーションの精度において、報告されていた通り MedSAM (平均 dice=0.8768) が SAM (平均 dice=0.9056) より優れていることを確認しました。結果を図5に示します。
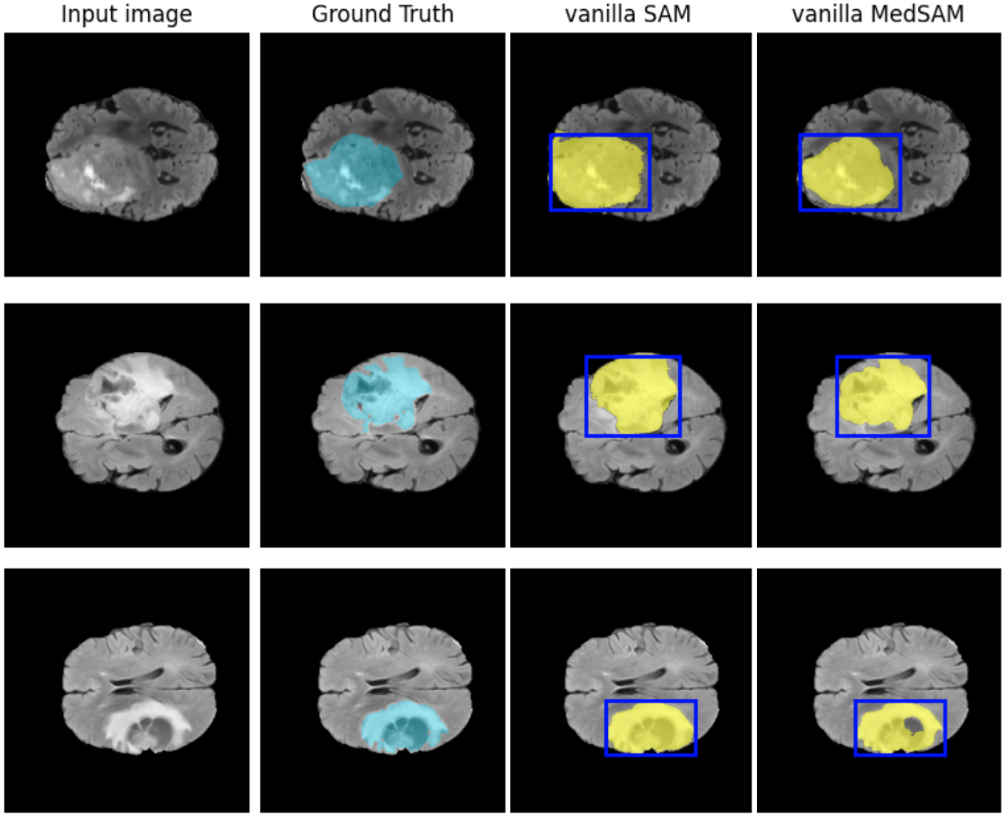
図4. SAM と MedSAM によるセグメンテーションの例
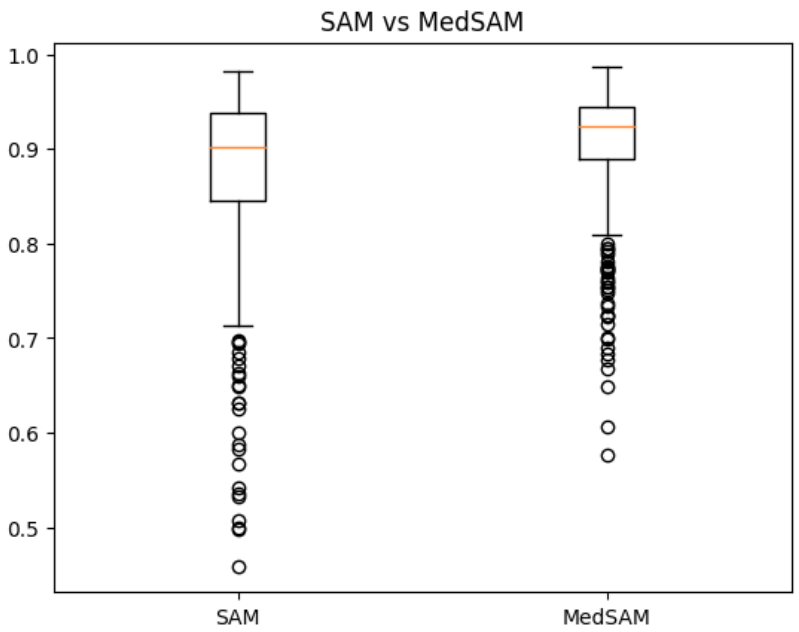
図5. SAM と MedSAM の精度比較
この結果を受けて、パラメータの初期値として MedSAM の学習済みパラメータを採用することとして、fine-tuning における追加学習を 200 epoch で行いました。また SAM のセグメンテーションはプロンプトに強く依存するため、全ての手法で bounding box のプロンプトを与えました。
Fine-tuning の手法についてはいくつかの条件で比較しました。本記事ではインターンで行った実験の一部を割愛して以下の4条件において比較を行います。Decoder-tuning では図1に示したアーキテクチャのうち Mask Decoder の全パラメータのみを学習可能なパラメータとします。Bias-tuning は AdaptiveSAM で用いられた fine-tuning 手法を指し、Mask Decoder だけでなく bias 項など複数種類のパラメータを更新します。すなわち、図1における Image Encoder や Prompt Encoder のパラメータの一部も更新される点が decoder-tuning と異なる点です。Non-decoder-tuning では、bias-tuning で更新されるパラメータのうち Mask Decoder のパラメータのみを学習の対象から除外しました。各 fine-tuning 手法において更新されるパラメータ数を図6に示します。
- vanilla MedSAM: fine-tuningなし
- decoder-tuning: Mask Decoder
- bias-tuning: Mask Decoder + norm + positional-embedding + prompt-encoder + bias
- non-decoder-tuning: norm + positional-embedding + prompt-encoder + bias
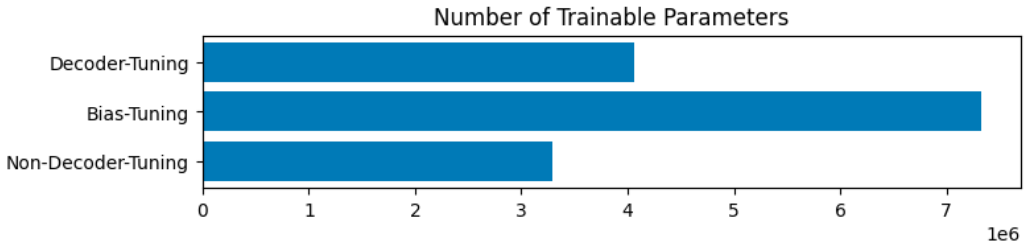
図6. 各fine-tuning手法における訓練可能なパラメータ数
MedSAM と各 fine-tuning 手法で訓練されたモデルの出力の例を図7に示します。MedSAM では Ground Truth の領域を塗りつぶすようなセグメンテーションマスクを出力しています。Decoder-tuning ではマスクの中央に穴が開きより Ground Truth に近い出力が得られており、bias-tuning や non-decoder-tuning ではさらに細部を正確に表現することに成功しています。
test データの平均 Dice スコアの比較を図8に示します。全ての手法で vanilla MedSAM より高いスコアが得られていますが、decoder-tuning では他の手法と比較して僅かに劣っています。
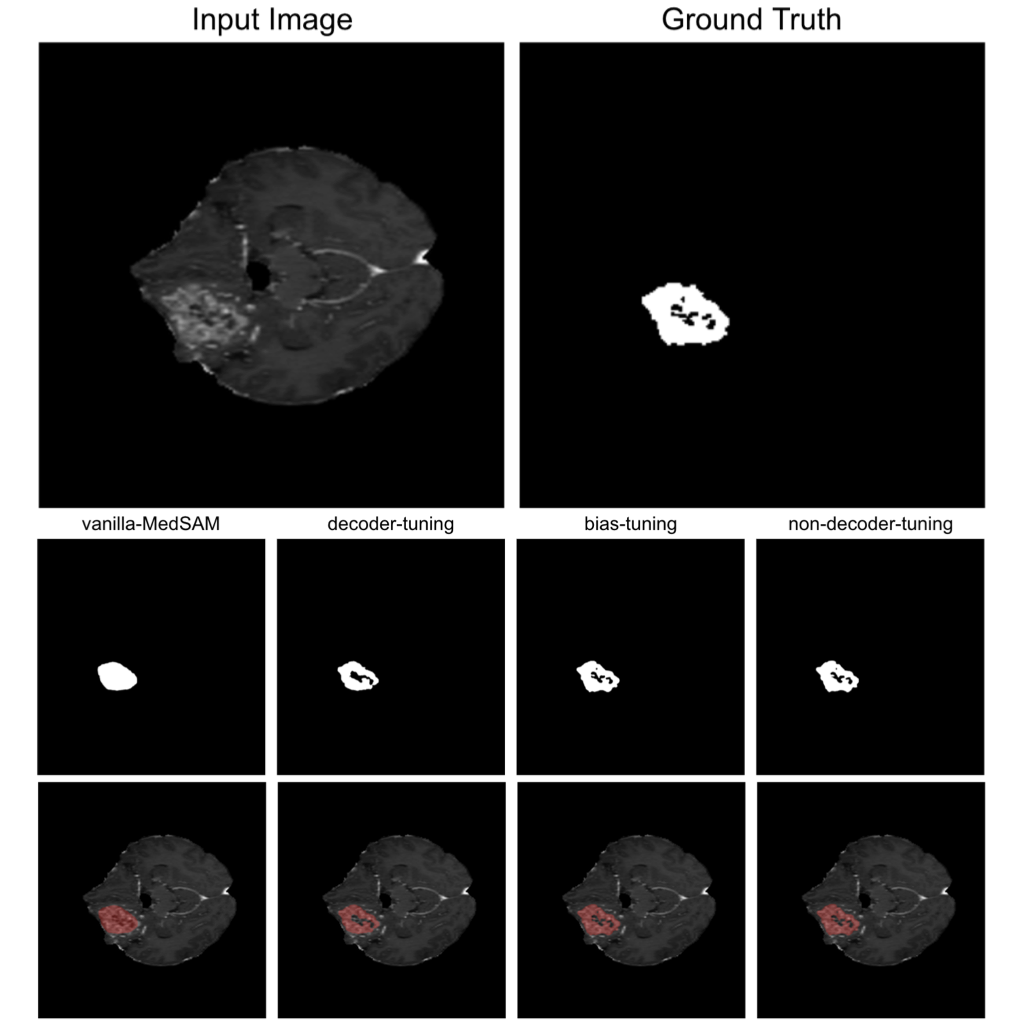
図7. MedSAM と各 fine-tuning モデルのセグメンテーションの例
各条件における dice スコアはそれぞれ vanilla MedSAM = 0.8432, decoder tuning = 0.8665, bias tuning = 0.9256, non-decoder-tuning = 0.9215 を示した。
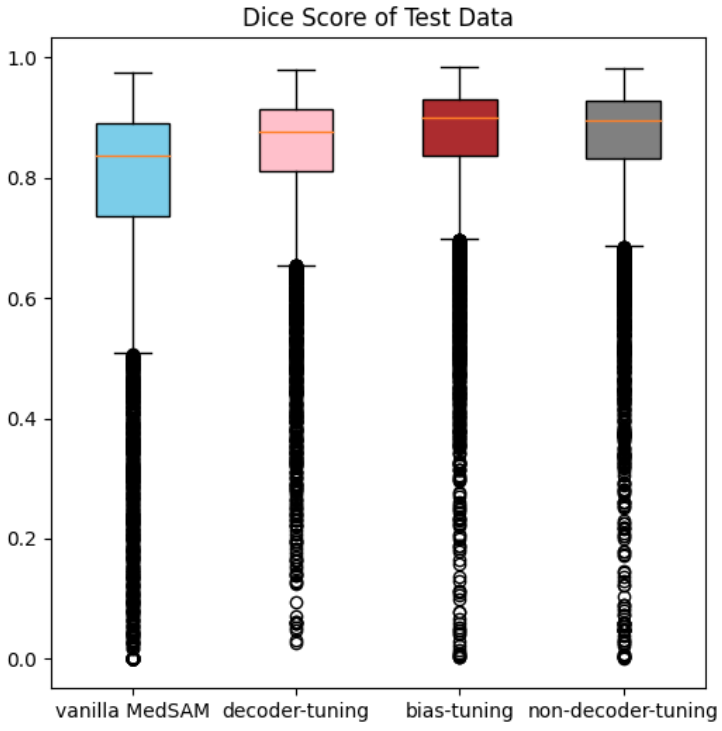
図8. MedSAM と各 fine-tuning モデルの精度比較
各条件における平均 dice スコアはそれぞれ vanilla MedSAM = 0.7699, decoder tuning = 0.8371, bias tuning = 0.8603, non-decoder-tuning = 0.8551 を示した。
さらに、図8に示したスコアを In-Distribution と OOD に分けて比較した結果を図9に示します。In-Distribution だけでなく OOD についても vanilla MedSAM よりスコアが上昇していることが分かります。
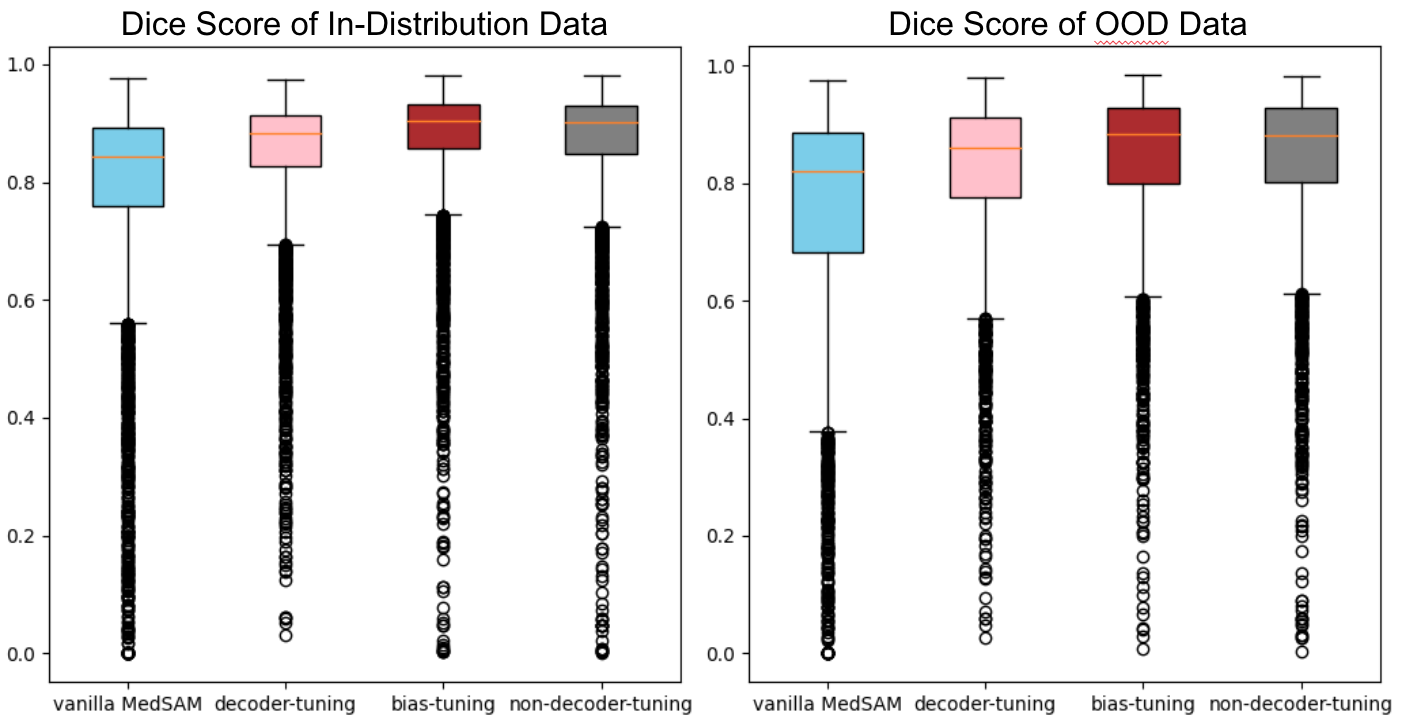
図9. In-Distribution データと OOD データにおけるモデル間比較
In-Distribution における各条件の平均 dice スコアはそれぞれ vanilla MedSAM = 0.7832, decoder tuning = 0.8490, bias tuning = 0.8721, non-decoder-tuning = 0.8666 を示した。また OOD における各条件の平均 dice スコアはそれぞれ vanilla MedSAM = 0.7444, decoder tuning = 0.8145, bias tuning = 0.8380, non-decoder-tuning = 0.8333 を示した。
さらに各 fine-tuning 条件における施設ごとの精度比較も行いました。図10,11および表1,2 に In-Distribution と OOD それぞれにおける施設ごとの比較を示します。
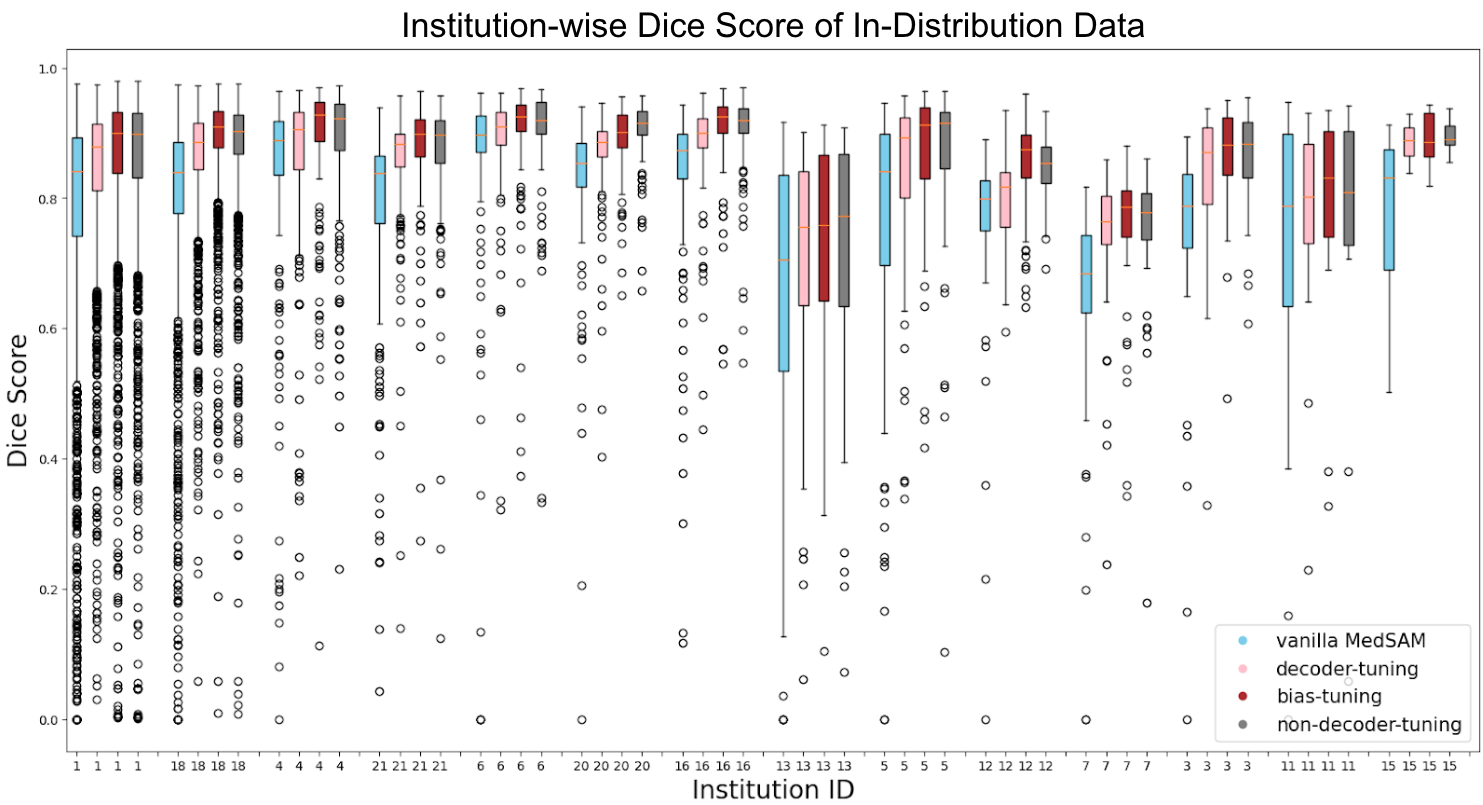
図10. In-Distribution の施設ごとのモデル間比較
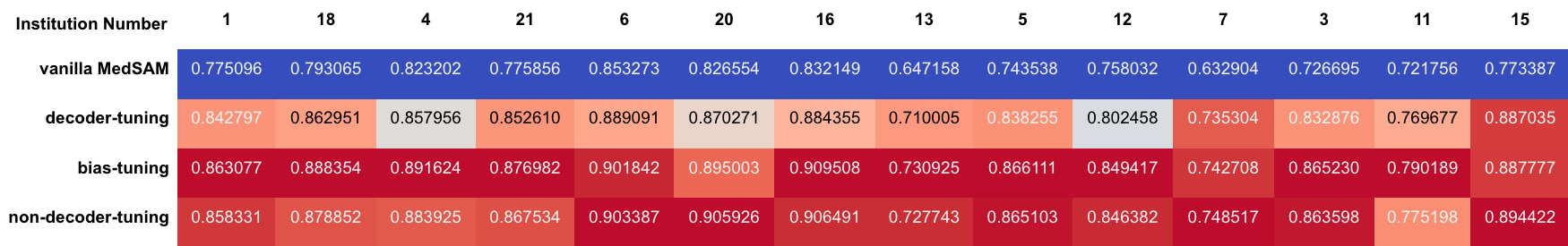
表1. In-Distribution の施設ごとの平均 Dice スコア
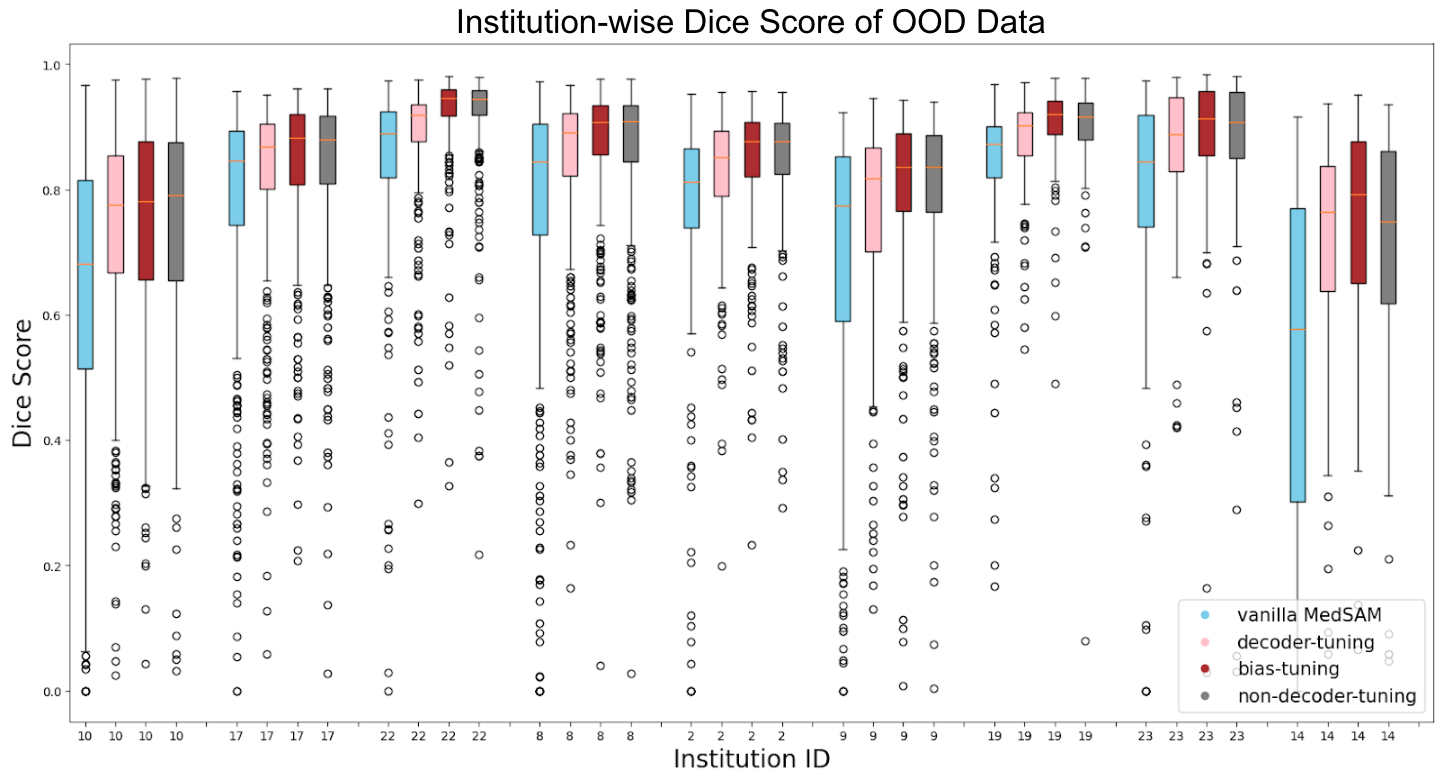
図11. Out-of-Distribution の施設ごとのモデル間比較
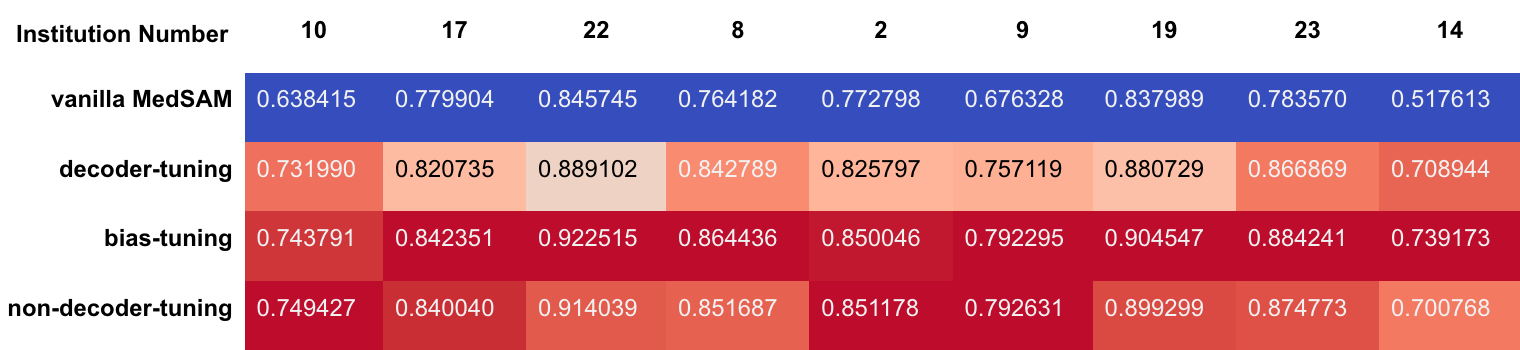
表2. Out-of-Distribution の施設ごとの平均 Dice スコア
図10, 11および表1, 2 から、全体的な傾向として bias-tuning のセグメンテーション精度がどの施設に対しても頑健であり、non-decoder-tuning のセグメンテーション精度も一部の施設を除いて次点で高いことがわかりました。一方で、図11の OOD 施設14などのように他の施設と比較して精度が低い施設が見られます。このような特定のケースに注目すると bias-tuning は他の手法よりスコアが高く、non-decoder-tuning はスコアが低い傾向にあります。このような傾向が生まれる仮説としては、図6に示したように fine-tuning において学習パラメータ数がより多いほど、vanilla MedSAM が元々苦手としていた症例に対してもセグメンテーション精度がより頑健になるということが考えられます。
Discussion
今回の fine-tuning 手法の比較から、MedSAM を起点として fine-tuning を行う際には、図8-10に示した結果から Decoder のみを学習パラメータとすると精度が上がりにくく、図10,11の施設ごとの結果から Decoder を学習対象から除外すると一部の症例に対し精度が低くなりやすいことが分かりました。したがって、Decoder に加えて bias-tuning のように Image Encoder などの一部のパラメータについても学習対象に含めることが有効だと考えられます。ただし、図10,11において見られた MedSAM のセグメンテーション精度が低い特定の施設の症例に対する頑健性と学習パラメータ数との関係や、特定のレイヤーが精度に影響を与えているかなどについては、今後より詳細な実験が必要です。
また、今回は多施設から医用画像データセットとしては比較的多くの症例を用いて学習を行いましたが、過去のインターン生の方が同じく FeTS2022 データセットを用いて検証しているように[6]、1施設のみの小規模データなどで学習した場合の OOD の影響などについても今後の検証が必要です。
そのほか、今回明確なデータは出していませんが、SAM はプロンプト入力の影響が大きいため、実験設定の中でどの程度正確なプロンプトを与えるかは結果に重要になるのではないかと考えています。Bounding box などのプロンプトを一切与えずに学習するなど、単純な手法でプロンプト非依存的なモデル構築を目指した場合には、特定の症例で急激にスコアが落ちるなど、精度の不安定化を招くのではないかという仮説を試行錯誤する中で抱きました。この点についても、まだまだ検証・改善の余地は大きいと思われます。
Appendix
本記事では、SAM の医用画像セグメンテーションのための fine-tuning 手法の比較検討について執筆させていただきましたが、インターンシップで取り組んだもうひとつのテーマとして『画像メタデータをプロンプトとした医用画像セグメンテーションの精度改善』がありました。ここでの画像メタデータとは CT や MRI のデータに付属した患者や撮像機器などの情報を指します。
近年、皮膚がんの画像診断などに対し、臨床メタデータ(疾患の統計的情報や患者の年齢・性別など)の活用が疾患の識別精度を改善するという趣旨の論文がいくつか報告されています [7]。しかし、セグメンテーションタスクにおける画像メタデータの適用例は著者が探す限りでは見つかっていません。臨床的に明確に有意義なデータに限らず、撮像設定などの情報を用いる事で、セグメンテーションの精度をロバストにできるのではないかという仮説を検証しました。その際に、AdaptiveSAM のテキストプロンプトの機構をメタデータの入力部分として転用しています。メタデータの有効性を検証するにあたり、The Lung Image Database Consortium and Image Database Resource Initiative (LIDC-IDRI) [8]というプロジェクトの中で作成された肺 CT のデータセットを用いました。結論としては、今回の期間中では残念ながらその有効性は示せませんでした。
ただし、図12に示すように個々の症例を見るとメタデータの有無による精度の変動は確認できるため、適切に有効な臨床情報や画像メタデータを取捨選択することが精度向上の決め手になるのではないかと考えています。
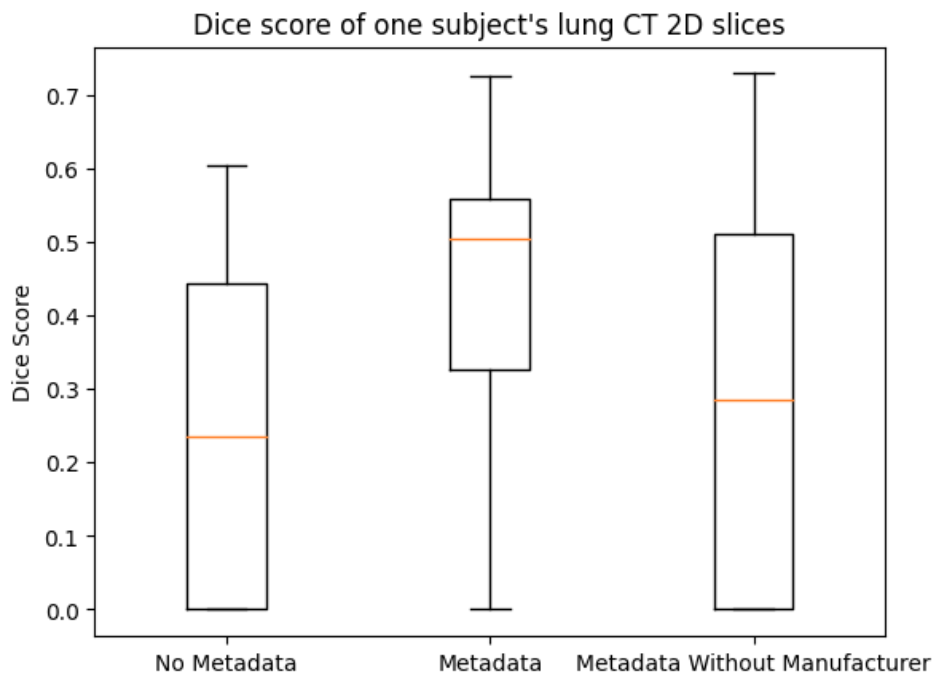
図12. メタデータの有無に伴う Dice スコアの変動の例
各箱ひげ図のうち、No Metadata はメタデータを用いずに bias-tuning を行ったモデルのDice スコアを表している。Metadata は複数項目のメタデータをプロンプトとして与えて bias-tuning を行い、テスト時に同一の項目のメタデータを与えている。Metadata Without Manufacturer は Metadataと同一の bias-tuning を行ったモデルに対し、テスト時のみ CT の製造メーカー名についての項目を除いたメタデータを与えて Dice スコアを計算している。
終わりに
これまで医学系の研究室で我流で機械学習に触れていた私にとって、今回のインターンシップは大変学びの多い機会となりました。特に、機械学習による実験の基礎の所から、足りない点を多々自覚し改善するきっかけをいただけたのは大きかったです。
至らない点も多い中、研究テーマの決定から、実験の進行、発表に至るまで毎日メンターのお二人および同チームの方々に親身にアドバイスをいただき、様々な知見を持つインターン同期との交流にも恵まれ、大変貴重な一ヶ月半でした。
お世話になりましたメンターの徳岡さん、小田さん、および Medical Image チームの皆さまに心より感謝申し上げます。
参考文献
- Alexander Kirillov et al. “Segment Anything”, arXiv preprint arXiv:2304.02643 (2023).
- Jun Ma, Yuting He, Feifei Li, Lin Han, Chenyu You, Bo Wang. “Segment Anything in Medical Images”, arXiv preprint arXiv:2304.12306 (2023).
- Jay N. Paranjape et al. “AdaptiveSAM: Towards Efficient Tuning of SAM for Surgical Scene Segmentation”, arXiv preprint arXiv:2308.03726 (2023).
- Elad Ben Zaken, Shauli Ravfogel, Yoav Goldberg. “BitFit: Simple Parameter-efficient Fine-tuning for Transformer-based Masked Language-models”, arXiv preprint arXiv:2106.10199 (2021)
- S.Pati, U.Baid, M.Zenk, B.Edwards, M.Sheller, G.A.Reina, et al., “The Federated Tumor Segmentation (FeTS) Challenge”, arXiv preprint arXiv:2105.05874 (2021).
- Zongyao Li and Junichiro Iwasawa. “Test-time adaptation for brain tumor segmentation with cross-institutional MRI”, Preferred Networks Research and Development 2022. https://tech.preferred.jp/en/blog/i22-test-time-adaptation-for-brain-tumor-segmentation/
- A. G. C. Pacheco and R. A. Krohling, “An Attention-Based Mechanism to Combine Images and Metadata in Deep Learning Models Applied to Skin Cancer Classification”, Sept. 2021, doi: 10.1109/JBHI.2021.3062002.
- Armato SG 3rd et al. “The Lung Image Database Consortium (LIDC) and Image Database Resource Initiative (IDRI): A completed reference database of lung nodules on CT scans”, Medical Physics, 38: 915–931, 2011. DOI: https://doi.org/10.1118/1.3528204